博文
计算进化树末端节点出现的时间及两节点共同祖先(MRCA)的年龄
|||
Jinlong Zhang
1/10/2020
目的:
建立进化树
获得进化树中每个种的延续时间
获得某两个种最近共同祖先(MRCA)距离现在的时间
代码
setwd("C:/Users/jlzhang/Desktop/age")
library(plantlist)
# devtools::install_github("helixcn/plantlist")## This is plantlist 0.6.1.
library(V.PhyloMaker)
# devtools::install_github("jinyizju/V.PhyloMaker")library(phytools) library(dispRity)
library(openxlsx) library(phytools)
用中文名建进化树
查询学名
注意xlsx中保存的是中文名
species <- read.xlsx("checklist.xlsx")
taxa.table(TPL(CTPL(species[,1])$SPECIES), "taxa_table.txt")## Warning in CTPL(species[, 1]): Taxa: 红枝柴 ## could not be recognized.
## Note: too many rows to show, only the first few rows were printedTAXA_NAME : ## [1] "木荷" "厚皮香" "狗骨柴" "杜鹃" "薄叶山矾" "赤楠" ## ## SPECIES : ## [1] "Schima superba" "Ternstroemia gymnanthera" ## [3] "Diplospora dubia" "Rhododendron simsii" ## [5] "Symplocos anomala" "Syzygium buxifolium" ## ## SPECIES_FULL : ## [1] "Schima superba Gardn. et Champ." ## [2] "Ternstroemia gymnanthera (Wight et Arn.) Beddome" ## [3] "Diplospora dubia (Lindl.) Masam." ## [4] "Rhododendron simsii Planch." ## [5] "Symplocos anomala Brand" ## [6] "Syzygium buxifolium Hook. et Arn." ## ## GENUS : ## [1] "Schima" "Ternstroemia" "Diplospora" "Rhododendron" "Symplocos" ## [6] "Syzygium" ## ## GENUS_AUTHOR : ## [1] "Reinw. ex Blume" "Mutis ex L. f." "DC." "L." ## [5] "Jacq." "Gaertn." ## ## GENUS_CN : ## [1] "木荷属" "厚皮香属" "狗骨柴属" "杜鹃花属" "山矾属" "蒲桃属" ## ## FAMILY : ## [1] "Theaceae" "Pentaphylacaceae" "Rubiaceae" "Ericaceae" ## [5] "Symplocaceae" "Myrtaceae" ## ## FAMILY_CN : ## [1] "山茶科" "五列木科" "茜草科" "杜鹃花科" "山矾科" "桃金娘科" ## ## FAMILY_NUMBER : ## [1] "APGIII_334" "APGIII_330" "APGIII_350" "APGIII_344" "APGIII_335" ## [6] "APGIII_222" ## ## ORDER : ## [1] "Ericales" "Ericales" "Gentianales" "Ericales" "Ericales" ## [6] "Myrtales" ## ## GROUP : ## [1] "Angiosperms" "Angiosperms" "Angiosperms" "Angiosperms" "Angiosperms" ## [6] "Angiosperms" ## ## IUCN_CHINA : ## [1] "无危(LC)" "无危(LC)" "无危(LC)" "无危(LC)" "无危(LC)" ## [6] "无危(LC)" ## ## ENDEMIC_TO_CHINA : ## [1] "否" "否" "否" "否" "否" "否" ## ## PROVINTIAL_DISTRIBUTION : ## [1] "浙、赣、湘、黔、闽、台、粤、桂、琼、港" ## [2] "皖、浙、赣、湘、鄂、川、黔、闽、粤、桂、滇、琼、港" ## [3] "皖、苏、浙、赣、湘、川、闽、台、粤、桂、滇、琼、港" ## [4] "皖、苏、浙、赣、湘、鄂、川、黔、闽、台、粤、桂、滇" ## [5] "皖、苏、浙、湘、鄂、川、黔、闽、台、粤、桂、滇、藏、琼" ## [6] "皖、浙、赣、湘、黔、闽、台、粤、桂" ## ## ALTITUDE : ## [1] "1000" "200-1400(2000-2800云南)" ## [3] "40-1500" "500-1200(-2500)" ## [5] "400-3000" ""
## [1] "Theaceae/Schima/Schima_superba" ## [2] "Pentaphylacaceae/Ternstroemia/Ternstroemia_gymnanthera" ## [3] "Pentaphylacaceae/Cleyera/Cleyera_japonica" ## [4] "Rubiaceae/Gardenia/Gardenia_jasminoides" ## [5] "Rubiaceae/Diplospora/Diplospora_dubia" ## [6] "Ericaceae/Rhododendron/Rhododendron_simsii" ## [7] "Symplocaceae/Symplocos/Symplocos_anomala" ## [8] "Symplocaceae/Symplocos/Symplocos_sumuntia" ## [9] "Symplocaceae/Symplocos/Symplocos_lucida" ## [10] "Symplocaceae/Symplocos/Symplocos_stellaris" ## [11] "Myrtaceae/Syzygium/Syzygium_buxifolium" ## [12] "Aquifoliaceae/Ilex/Ilex_ficoidea" ## [13] "Aquifoliaceae/Ilex/Ilex_rotunda" ## [14] "Aquifoliaceae/Ilex/Ilex_micrococca" ## [15] "Rutaceae/Evodia/Evodia_glabrifolia" ## [16] "Daphniphyllaceae/Daphniphyllum/Daphniphyllum_oldhami" ## [17] "Lauraceae/Litsea/Litsea_elongata" ## [18] "Lauraceae/Litsea/Litsea_cubeba" ## [19] "Rosaceae/Photinia/Photinia_beauverdiana" ## [20] "Moraceae/Ficus/Ficus_erecta" ## [21] "Myricaceae/Myrica/Myrica_rubra" ## [22] "Styracaceae/Styrax/Styrax_suberifolius" ## [23] "Fagaceae/Castanopsis/Castanopsis_carlesii" ## [24] "Fagaceae/Cyclobalanopsis/Cyclobalanopsis_gracilis" ## [25] "Ebenaceae/Diospyros/Diospyros_morrisiana" ## [26] "Elaeocarpaceae/Elaeocarpus/Elaeocarpus_japonicus" ## [27] "Betulaceae/Carpinus/Carpinus_viminea" ## [28] "Pittosporaceae/Pittosporum/Pittosporum_illicioides"
读取查询到的学名
tab <- read.table("taxa_table.txt", sep = "/")
colnames(tab) <- c("family", "genus", "species")
tab2 <- tab[, c("species", "genus", "family")]用V.PhyloMaker建树,速度较慢
result1 <- phylo.maker(tab2, scenarios = c("S1"))
write.tree(result1[[1]], "tree1.newick")
# 保存到硬盘,用于用其他软件(如Figtree)查看
tree <- result1[[1]]获得每个种延续的时间
注意,这个与进化树收录的分类单元密切相关,一般不宜用于研究。
脚本来源 http://blog.phytools.org/2013/10/finding-edge-lengths-of-all-terminal.html
n <- length(tree$tip.label) ee <- setNames(tree$edge.length[sapply(1:n, function(x, y) which(y == x), y = tree$edge[, 2])], tree$tip.label) ee <- as.data.frame(ee) write.xlsx(ee, "terminal_branch_length.xlsx", row.names = TRUE)
查询两个分类单元最近共同祖先的节点名
tree$node.label <- paste(1:tree$Nnode + Ntip(tree))
name_node <- getMRCA(tree, tip = c("Cleyera_japonica", "Rhododendron_simsii"))
name_node_ind <- tree$node.label[name_node - Ntip(tree)]绘制进化树
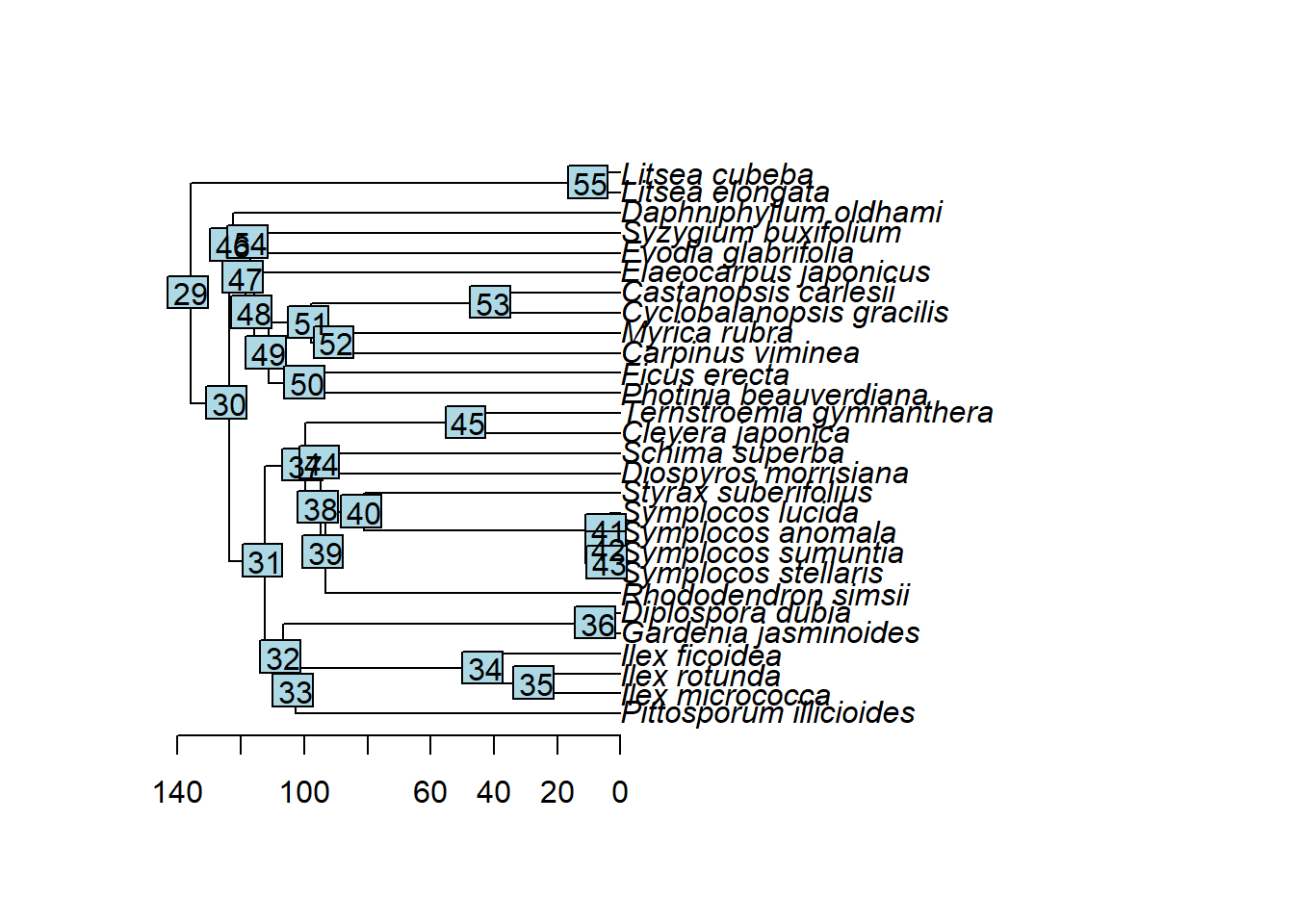
plot(tree) axisPhylo(1, las = 1) ## see nodelabels(text=tree$node.label, node=1:tree$Nnode+Ntip(tree))
plot(tree)
axisPhylo(1, las = 1)
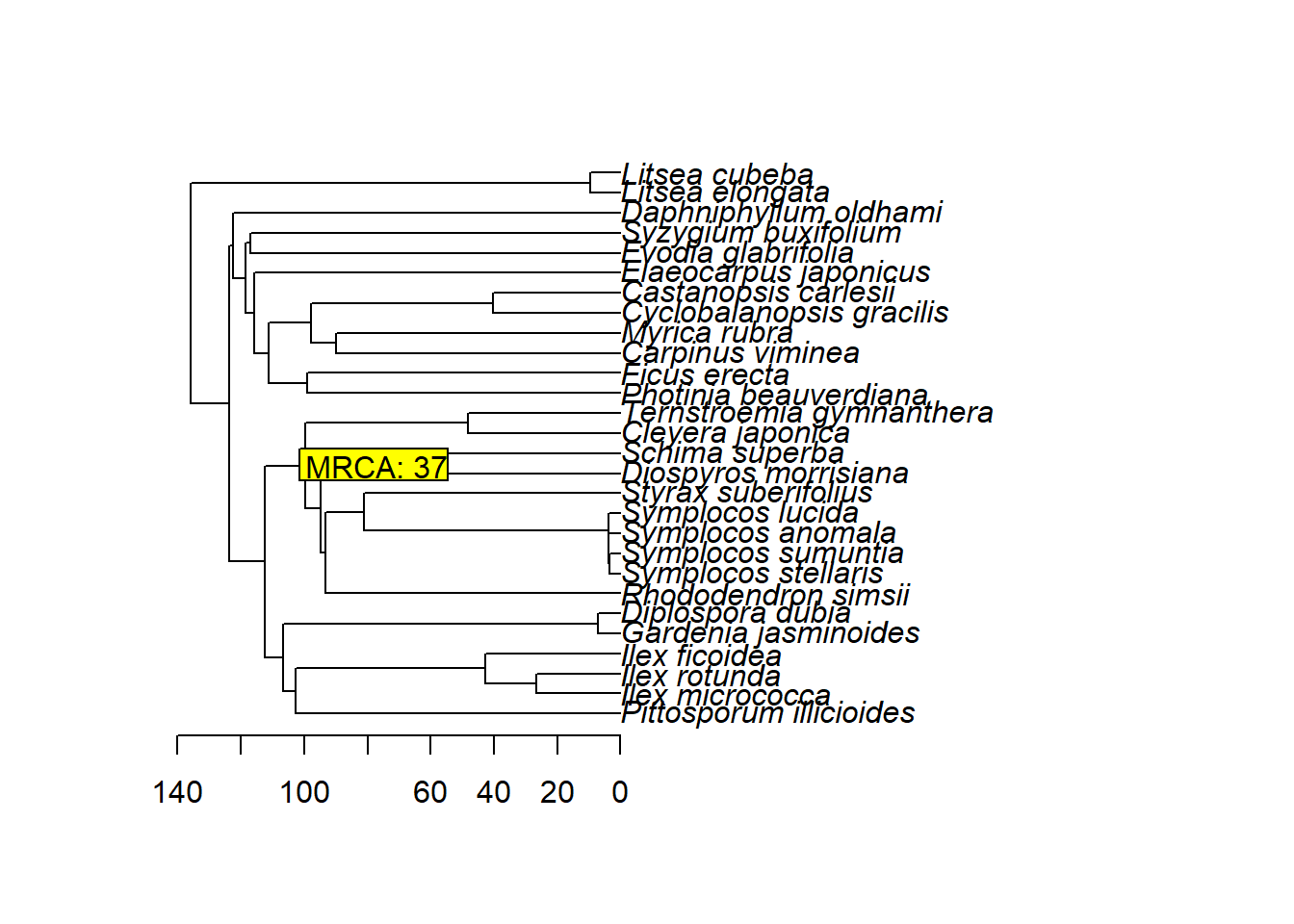
nodelabels(paste("MRCA:", name_node), name_node, frame = "r", bg = "yellow", adj = 0)查询两个种的最近共同祖先MRCA的时间
### From the dispRity Package age_dat <- tree.age(tree) age_dat[as.character(age_dat$elements) == name_node_ind, ]
## ages elements ## 37 99.706 37
更多内容
ggtree用于进化树的绘制 https://yulab-smu.github.io/treedata-book/chapter5.html#visualizing-and-annotating-tree-using-grammar-of-graphics
https://m.sciencenet.cn/blog-255662-1213612.html
上一篇:用phylotools建立supermatrix
下一篇:再忆铁峰