博文
PCNM动画展示:生态学与空间统计学
||||
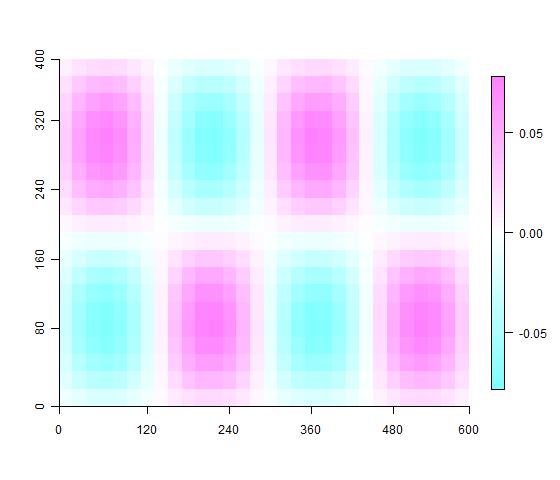
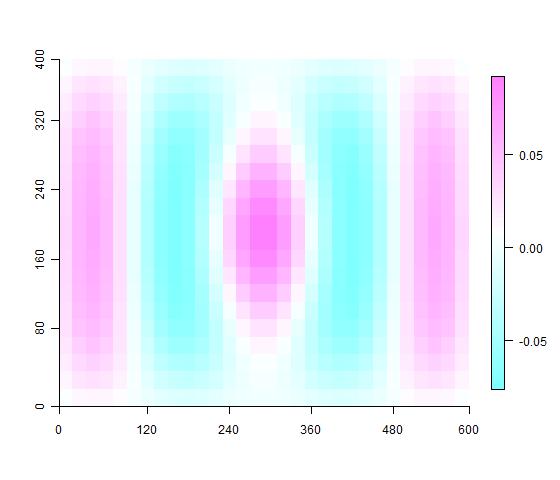
PCNM (Principal Coordinates of Neighbourhood Matrix)是Legendre教授等开发出的表示空间关系的方法,类似于pcoa。常用于生物多样性空间格局研究中,例如在古田山beta多样性研究中,Legendre(2009)就使用了这种方法。用样地的规则栅格距离可以对点两两之间的距离关系进行PCNM模拟。 这里用到了vegan程序包将距离矩阵转换成PCNM的特征向量,用imagevect函数绘制PCNM特征向量在样地中的分布。希望对有兴趣的研究人员有帮助。
将以下代码在R中运行即可。
#### Function imagevect
#### By Jinlong Zhang <Jinlongzhang01@gmail.com>
#### Institute of Botany, the Chinese Academy of Sciences, Beijing ,China
#### Aug. 18, 2011
imagevect <- function (x, labels, contour = FALSE, gridsize = 20,
axes = TRUE, nlabx = 5, nlaby = 5, ...)
{
require(fields)
dimension <- function(x, unique = FALSE, sort = FALSE){
ncharX <- substring(x, 2, regexpr("Y", x)-1)
ncharY <- substring(x, nchar(ncharX)+3, nchar(x))
if(unique){
ncharX = unique(ncharX)
ncharY = unique(ncharY)
}
if(sort){
ncharX = sort(as.numeric(ncharX))
ncharY = sort(as.numeric(ncharY))
}
res <- list(ncharX, ncharY)
return(res)
}
formatXY <- function(x){
ncharX <- substring(x, 2, regexpr("Y", x)-1)
ncharY <- substring(x, nchar(ncharX)+3, nchar(x))
resX <- c()
for(i in 1:length(ncharX)){
n0x <- paste( rep(rep(0, length(ncharX[i])),
times = (max(nchar(ncharX)) - nchar(ncharX[i]) + 1)),
collapse = "", sep = "")
resX[i] <- paste("X", substring(n0x, 2, nchar(n0x)), ncharX[i], collapse = "", sep = "")
}
resY <- c()
for(i in 1:length(ncharY)){
n0x <- paste( rep(rep(0, length(ncharY[i])),
times = (max(nchar(ncharY)) - nchar(ncharY[i]) + 1)),
collapse = "", sep = "")
resY[i] <- paste("Y", substring(n0x, 2, nchar(n0x)), ncharY[i], collapse = "", sep = "")
}
res <- paste(resX, resY, sep = "")
return(res)
}
sort.x <- x[order(formatXY(labels))]
rrr <- dimension(labels, unique = TRUE, sort = TRUE)
dims <- c(length(rrr[[2]]), length(rrr[[1]]))
dim(sort.x) <- dims
par(xaxs = "i", yaxs = "i")
image.plot(nnn <- t(sort.x), axes = FALSE, ...)
if (contour) {
contour(nnn, add = TRUE, ...)
}
if (axes) {
points(0, 0, pch = " ", cex = 3) ## invoke the large plot
get.axis.ticks <-
function(nlabs = NULL, gridsize = NULL, limit_max = NULL){
ngrid <- (limit_max-0)/gridsize ## Obtain number of labels to plot
per_grid <- 1/(ngrid-1) ## Obtain length for each grid
start <- 0 - (1/(ngrid-1))/2 ## starting point for the ticks
stop <- 1 + (1/(ngrid-1))/2 ## stopping point for the ticks
lab <-(0:nlabs*(limit_max/nlabs))
at <- seq(from = start, to = stop,
by = ((1 + per_grid))/((length(lab)-1))) ## Position of the ticks
return(list(lab, at))
}
xaxis.position <- get.axis.ticks(nlabs = nlabx, gridsize = gridsize,
limit_max = gridsize * nrow(nnn))
yaxis.position <- get.axis.ticks(nlabs = nlaby, gridsize = gridsize,
limit_max = gridsize * ncol(nnn))
axis(1, labels = xaxis.position[[1]], at = xaxis.position[[2]])
axis(2, labels = yaxis.position[[1]], at = yaxis.position[[2]])
}
invisible(nnn)
}
library(vegan)
## 模拟一套数据
X <- seq(10, 590, by = 20)
Y <- seq(10, 390, by = 20)
## Label
XY <- expand.grid(X, Y)
names <- paste("X", (XY[,1] + 10)/20 ,"Y", (XY[,2] + 10)/20 , sep = "")
rownames(XY) <- names
## 计算样方两两之间的距离
distXY <- dist(XY)
## 进行pcnm
gtsPCNM <- pcnm(distXY)
head(gtsPCNM$vectors)
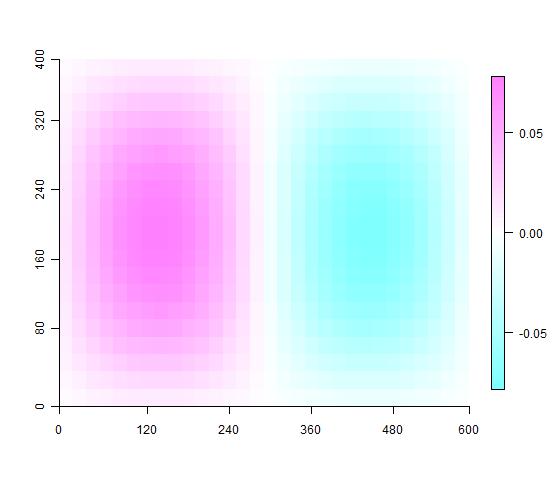
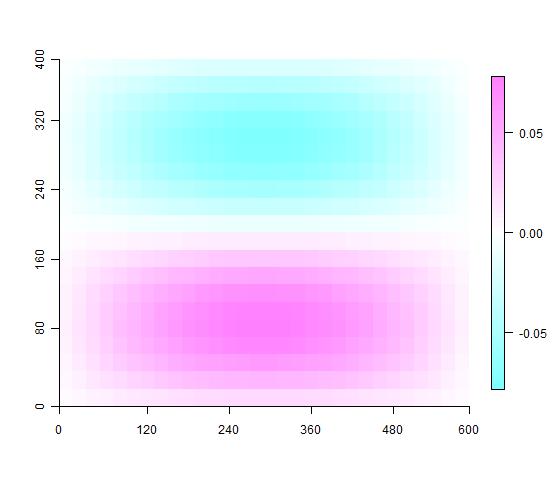
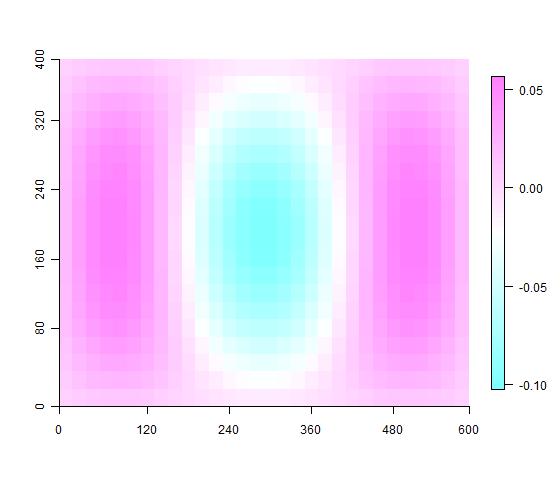
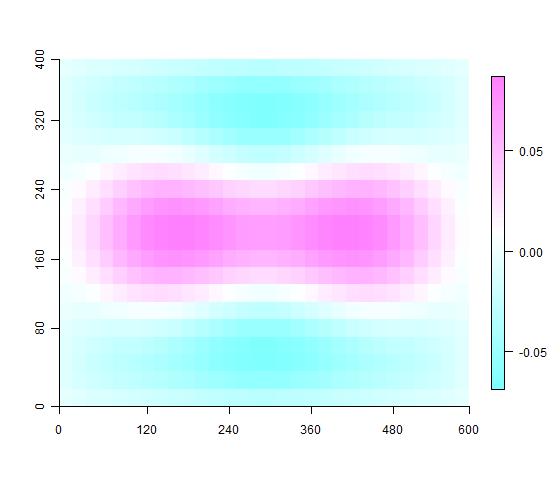
## 绘制pcnm图
for(i in 1:10){
imagevect(gtsPCNM$vectors[,i], labels = names,
col = topo.colors(100))
Sys.sleep(0.3)
}




https://m.sciencenet.cn/blog-255662-469451.html
上一篇:Tortoise SVN管理本地代码
下一篇:如何撰写生态学科研论文?