博文
Discovery Studio官方教程(Help-Tutorials) 分子动力学模拟
|
目的:采用Discovery Studio,以一组蛋白-配体复合物为实例,示范分子动力学及结果分析操作过程。
所需功能和模块:Discovery Studio Visualizer client, CHARMm, Simulation protocols
所需数据文件:1sem.pdb
所需时间:1小时
介绍
分子动力学(Molecular Dynamics, MD)是分子模拟中最常用的方法之一。该方法基于分子力场,能够动态的描述分子的运动状况,继而描述生命的动态过程。分子动力学在生命科学领域中的应用非常广泛,如蛋白质折叠的机理研究、酶催化反应的机理研究、与功能相关蛋白质的运动研究、生物大分子大范围构象变化的研究等等。近几十年来,分子动力学方法已经成功的运用于大分子体系低能量构象的模建、X射线晶体衍射以及NMR实验结果的处理。[1,2] 现在分子动力学方法已经成了理论生物学研究中必不可少的方法之一。[3,4]
分子动力学模拟主要包括如下几个步骤:
1. 模拟体系升温过程(Heating Stage)
2. 模拟体系平衡过程(Equilibrium Stage)
3. 模拟体系采样过程(Production Stage)
4. 分析体系的目标性质(Analysis)
然而,由于一般的待模拟体系的初始结构或多或少的存在缺陷(比如,初始结构不完整或存在不合理的结构区域),所以我们往往需要对初始结构进行预处理才能进行分子动力学模拟。一个完整的分子动力学模拟过程应包括如下几个步骤:
1. 初始结构检查及预处理
2. 给模拟体系赋力场参数
3. 考虑溶剂效应
4. 初始结构能量最小化
5. 动力学模拟(包括升温、平衡、采样)
6. 结果分析
目前,常用的分子动力学模拟软件都基于Unix(Linux)操作系统。实施每一个模拟的步骤都需要特定的命令来调用相关的程序对模拟体系进行处理。同时,模拟者还需要熟知每个参数的意义并定义相关的参数值。最终分析过程也需要配合其他软件才能完成。这对初级分子动力学模拟者而言是非常困难的(模拟者不仅需要掌握分子动力学软件的使用命令,还需要掌握操作系统相关的命令)。为了解决这样的问题,Discovery Studio为用户提供了基于窗口的分子动力学模拟工具。使用DS,用户可以轻松的完成分子动力学的整个模拟过程(包括体系的准备、模拟过程的设置及结果的分析)。另外考虑到初级模拟者在整个模拟流程方面不熟悉,DS专门设置了一个“Standard Dynamics Cascade”protocol。该Protocol将整个动力学模拟的步骤集合到一个参数设置界面当中,方便初级模拟者的使用。
为了使大家对MD的操作流程有所了解,本教程主要内容包括如下几个方面:
1. 初始结构的准备,力场设置
2. 对结构进行溶剂化处理
3. 标准的分子动力学模拟流程
4. 动力学模拟结果的轨迹分析
准备分子动力学体系,执行分子动力学计算
1. 蛋白结构预处理
双击图标,启动Discovery Studio,在文件浏览器(Files Explorer)双击打开Smaples | Tutorials | Simulation | 1sem.pdb文件,将蛋白-配体复合物导入3D窗口。(图1)

展开Marcomolecules | Protein Report工具面板,单击Protein Report。
Protein Report是蛋白质结构的重要信息报告。在报告中不仅提供了结晶的分辨率、分子量、氨基酸链名称/数量、实验pH值等基本信息。还提供了缺失或不完整的氨基酸残基会以洋红色的颜色标注出来。为后续结构修正和优化提供参考。
可以看出蛋白质复合物有两个相同的亚基组成,下一步我们需要判断保留哪个亚基进行随后的动力学模拟。
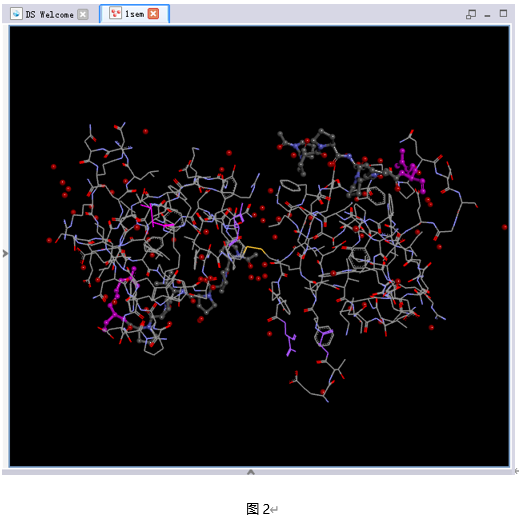
点击1sem标签,确保其处于激活状态。然后点击标准工具栏(Standard toolbar)中Display Style的下拉箭头,选择右下角的显示方式(蛋白质以线状表示,小分子以球棍状表示)。(图2)
再次单击Display Style,将Color by改为Isotropic Displacement。单击Colors…按钮,将颜色改为Blue-White-Red。图3
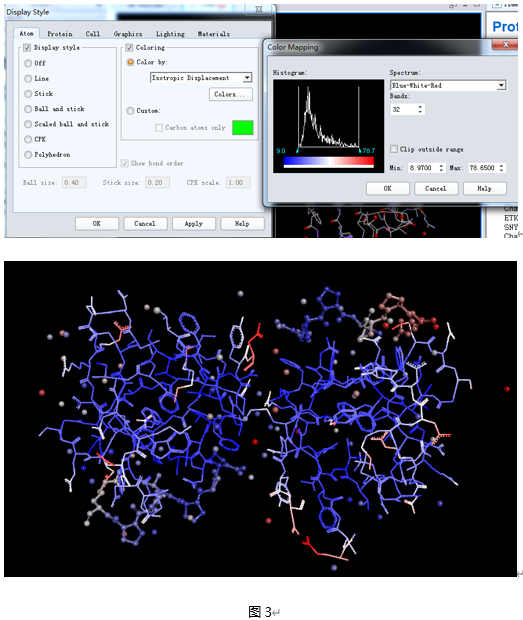
根据Isotropic displacement(也被称为B factor或温度因素)的数值,我们能够清晰的识别出每个原子位置的不确定性。值越大(红色)表示原子的位置变化越大。值越小(蓝色)热振动越小,位置越精确。
我们将C、D链进行对比,显然C链的红色更多。而D链基本为蓝色。另外根据Protein Report显示两个亚基的氨基酸残基的缺失或不完整的情况均较好。因此综合考虑后,保留B、D链。
快捷键CTRL+H,打开层级窗口(Hierarchy View),然后选择所有A、C链,快捷键delete删除。
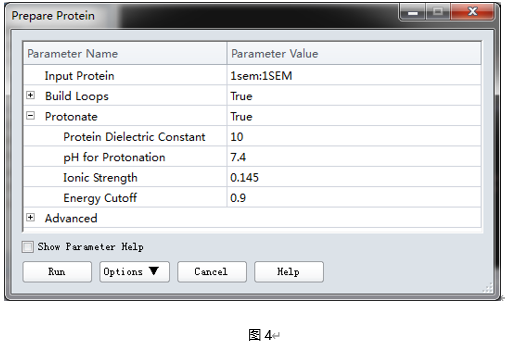
展开Macromolecules | Prepare Protein工具面板(Tool Panel),单击Prepare Protein....参数默认,点击run。等待任务完成。运行大约半分钟(图4)
1溶剂化蛋白质
打开处理过的1SEM_Prep文件,首先添加力场:打开Simulation工具面板,选择change forcefield,将Forcefield改为charmm36,然后再点击“Apply forcefield”。
从Protocol面板组中找到Simulation | Run Simulation文件夹,点击Solvation流程(图5),该流程在参数浏览器(Parameters Explorer)中打开,Input Typed Molecular输入1SEM_prep:1SEM,其它参数使用默认值,然后点击Run。
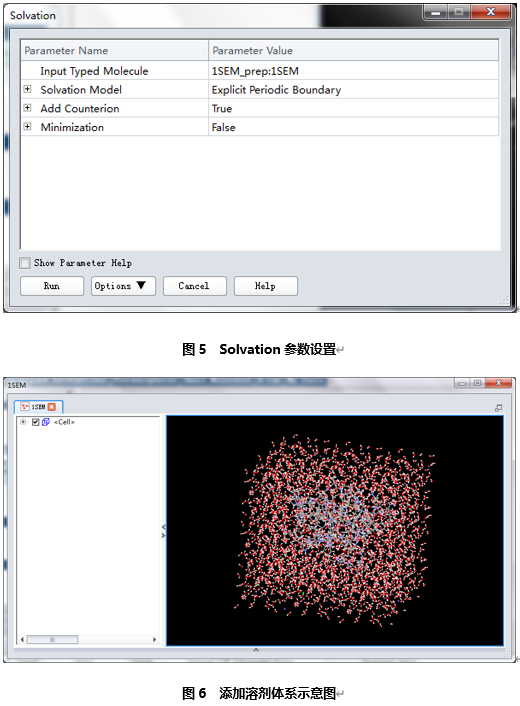
2打开动力学流程Standard Dynamics Cascade,修改参数
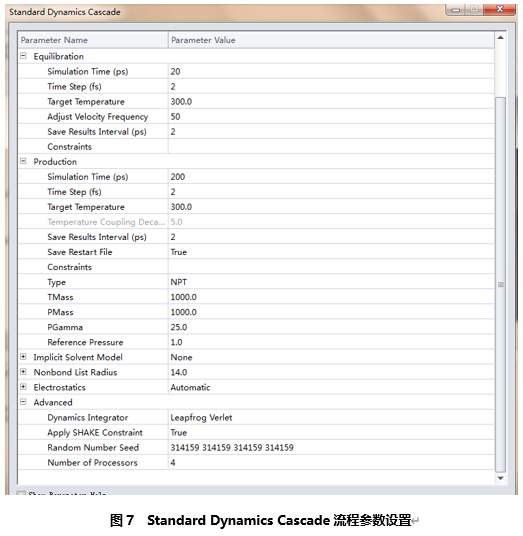
确保添加了溶剂体系的complex.dsv窗口为当前活跃窗口,文件流程浏览器(Protocol Explorer)中双击Simulations文件夹下dynamics文件夹下Standard Dynamics Cascade流程,该流程在参数浏览器(Parameters Explorer)中打开,“1SEM:1SEM”自动载入到Input Typed Molecule参数格。
Standard Dynamics Cascade流程包括5个阶段:优化I(Minimization)、优化II(Minimization2)、升温(Heating)、平衡(Equilibration)、模拟采样(Production)。
展开Equilibration阶段的参数组,设置Simulation Time(ps)为20。
展开Prodution阶段参数组,设置Simulation Time(ps)为200(图8)
展开Advanced,设置Number of Processors为8,其它参数保持默认,点击Run。
8核计算时,该作业需要2小时完成,作业的状态可以在“Jobs Explorer”中查看。当任务完成对话框打开,点击OK关闭。
3查看结果,显示构象变化动画
在Report页面,点击View Result链接。如果您没有运行完上述长时间的动力学模拟,也可以打开Samples | Tutorials | Simulation | 1SEM_nosolvent.dsv文件
选择下拉菜单Structure | Animation | Play命令,则可以看到在3D窗口中的复合物开始运动,显示动力学模拟过程中的变化。动画循环往复播放。再次选择下拉菜单Structure | Animation | Play命令可以停止动画播放。
分析分子动力学计算结果
您将通过Analyze Trajectory方法分析分子动力学轨迹的一些结构信息。该方法能够计算一些几何性质,如:距离、角度、二面角、或者非键相互作用的数量。另外也能够计算RMSD、RMSF。
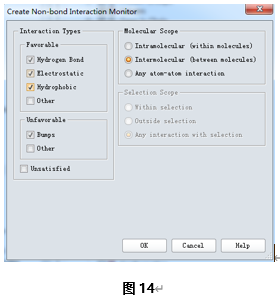
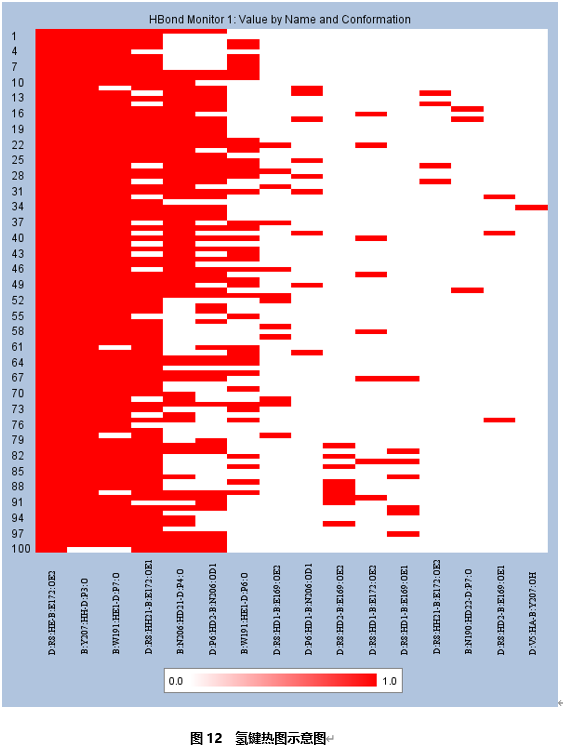
1定义非键相互作用监控
打开层级窗口(Hierarchy View),选择D链,然后点击标准工具栏的Non-bond Interactions… ,打开一个创建非键相互作用对话框。将Molecular Scope改为Intermolecular(between molecules),点击OK(图14)
,打开一个创建非键相互作用对话框。将Molecular Scope改为Intermolecular(between molecules),点击OK(图14)
2建立RMSD、RMSF,及编译动力学轨迹监控数据
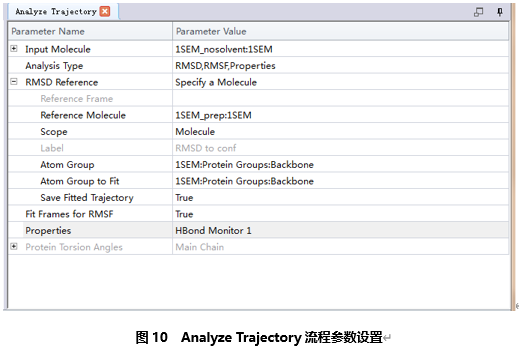
在流程浏览器(Tools Explorer)中展开Tools | Analyze Trajectory文件夹,单击下方的Analyze Trajectory...流程打开。
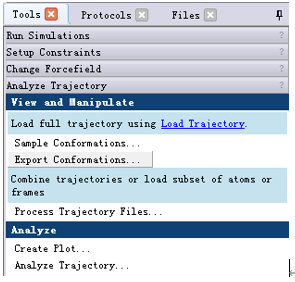
Input Molecule输入1SEM_nosolvent:1SEM
Analysis Type输入RMSD、RMSF、Properties。
点击RMSD Reference参数,下拉列表中选择Specify a Molecule。
展开RMSD Reference参数组,Reference Molecule 输入1SEM_prep :1SEM,确保Scope参数设置为Molecule。设置Atom Group及Atom Group to Fit参数为Backbone。
点击运行。
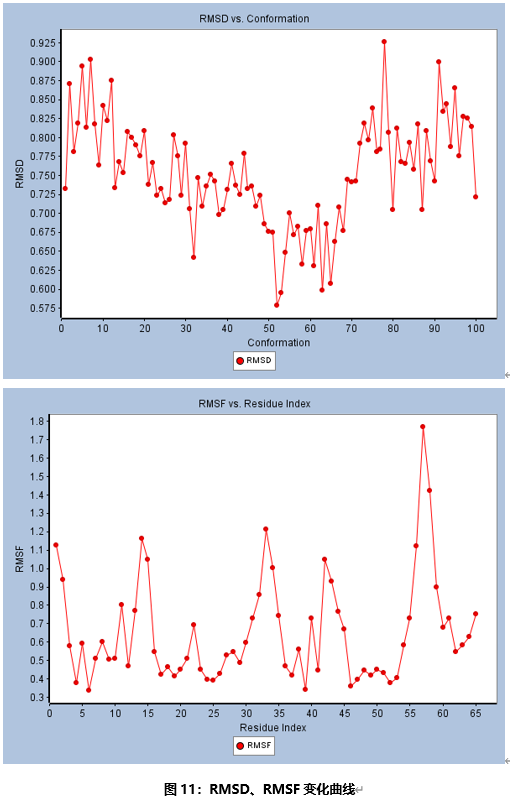
双击该作业行打开Output文件夹和Report.htm文件。点击打开View Results。

https://m.sciencenet.cn/blog-3536821-1362265.html
上一篇:Discovery Studio官方教程(Help-Tutorials) 使用MMPs识别生物活性(二)
下一篇:Discovery Studio官方教程(Help-Tutorials) 使用MCSS进行基于片段的分子对接