博文
Discovery Studio官方教程(Help-Tutorials) 药物设计之片段生长
|
目的:采用Grow Scoffold,在蛋白活性位点内从骨架进行先导化合物优化
所需功能和模块:Discovery Studio Client, DS CHARMM Lite, and DS CATALYST CONFORMATION.
所需数据文件:1kv2_complex.dsv, 2yis_scaffold.sd, diverse_sulfides.sd, sulfide_halfreaction.rxn.
所需时间:30分钟
介绍
先导化合物优化是一个复杂过程,为了得到一个临床前候选药物,通常需要对有前景化合物及骨架不断地进行化学结构优化,以提高化合物的活性、选择性、生物利用度、药效及药代动力学性质,并降低毒性。该过程通常是药物开发过程中的瓶颈。通过使用实用且有效的先导化合物优化软件,根据可获取的化合物试剂的快速推荐出容易合成的候选化合物的结构。基于结构的先导化合物优化主要集中于蛋白靶点的活性位点的化合物结构的设计。Grow Scaffold工具可以根据蛋白靶点活性位点的特点,通过基于化学反应的原位生长(reaction-based in situ enumeration)方法来找出那些能够产生潜在化合物的试剂,并对它们进行打分排序。
p38α是一个典型的丝氨酸/苏氨酸蛋白激酶,属于丝分裂原活化蛋白激酶(mitogen activated protein kinase)家族。它在内皮、免疫和炎性细胞中广泛表达,在肿瘤坏死因子和白细胞介素- 1等促炎症细胞因子产生的调控中扮演着重要作用。实验已经证明,选择性地抑制其中任何一个细胞因子都能够有效治疗各种炎症和免疫疾病,如风湿性关节炎、炎症性肠病,败血性休克,和骨质疏松症。二芳基脲是p38α的一个先导化合物
BoehringerIngelheim首先对它开展了先导化合物优化,随后Pfizer也开展了相关研究(图1)。
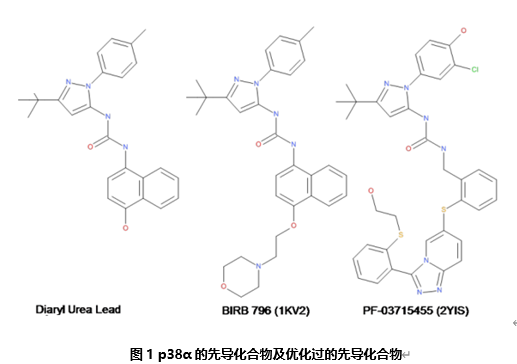
在本教程中你将使用Grow Scaffold流程来重现BoehringerIngelheim及Pfizer通过结构优化所发现的化合物。
本教程分为两部分:
(一)使用基于反应的原位生长方法产生优化过的先导化合物
1.执行优化计算
复合物结构采用PDB号为1KV2的晶体结构,本教程中所采用的该结构文件已经从PDB库中下载并经过Prepare Protein模块处理。
在文件浏览器(File explorer)中找到并双击打开Samples | Tutorials | Receptor-Ligand Interactions | 1kv2_complex.dsv.
该蛋白将在一个新的分子窗口中出现。与1KV2结合的配体(1kv2_xrayligand)是BIRB 796,它是第一报道的的p38αII型抑制剂。在本教程中,我们将在二芳基脲的萘环上引入一个吗啉乙氧基得到BIRB 796。
展开窗口左侧系统视图(Hierarchy View)(图2),勾掉1kv2_xrayligand复选框并选择1kv2_scaffold.选中脲基对位上萘基上的氢(H31)。
在工具浏览器(Tools Explorer)中,打开Receptor-Ligand Interactions | Lead Optimization工具栏,点击Grow Scaffold...打开 protocol对话框。
也可以在流程浏览器(Protocl explorer)中的Recepotor-LiandInteractions|Fragment Based Design|Grow Scaffold打开protocl。
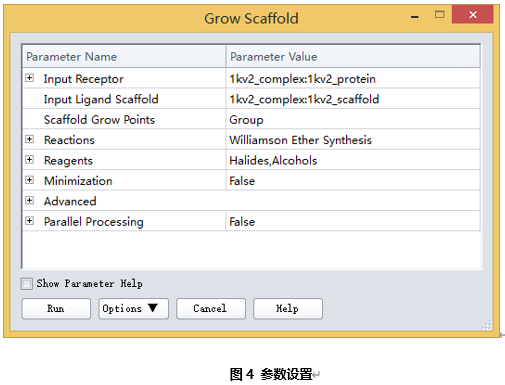
在参数浏览器中,点击Input Receptor参数,从下拉列表中选择1kv2_complex:1kv2_protein。
设置Input Ligand Scaffold参数为1kv2_complex:1kv2_scaffold。
点击Scaffold Grow Points选项,在下拉菜单中选择Create New Group From Selection..
点击Reactions参数,勾掉Amide Synthesis选项并选择Williamson Ether Synthesis选项。
点击Reagents参数,勾选Halides和Alcohols。
其他参数为默认,点击Run按钮运行。可以点击Background让作业后台运行,等待作业结束。
2.查看计算结果
点击作业浏览器(Job explorer)中刚完成的作业,点击View Results链接。
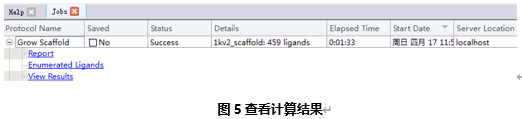
在打开的新的分子窗口1kv2_protein中确保系统视图和表格视图打开(可以分别使用快捷键Crtl+H和Crtl+T进行开关),在系统视图和表格视图中都可以观察到该窗口中包含先导化合物优化得到的444个分子。
点击表格视图中的 键和
键和 键,观察所产生的配体分子同受体分子的结合模式。
键,观察所产生的配体分子同受体分子的结合模式。
3.分析优化结果
非键相互作用的直观显示与分析
在工具浏览器(Tools Explorers)中,展开Receptor-Ligand Interactions | View Interactions,点击Ligand Interactions。
在视图窗口中,受体原子与所产配体间的非键相互作用会通过不同颜色的虚线显示出来,且只有参与了同配体之间的相互作用的残基才会显示。(图6)

点击上述View Interaction工具面板下的 按钮可以观察优化后的分子同蛋白之间的各种非键相互作用。
按钮可以观察优化后的分子同蛋白之间的各种非键相互作用。
为了更好的观察受体分子与配体对接pose间的相互作用,可以对体系进行旋转以获得最佳的观赏角度。
化合物的Pareto sorting方法进行排序,主要指标有:与蛋白形成的相互作用数目(最大化),违法lipiskk五规则数目(最小化),与受体发生碰撞数(最小化),片段的新颖性(最大化)的数量. 数据列表中也显示了配体与蛋白质间的相互作用,包括相互作用类型,蛋白质残基,作用距离等。
双击表格视图中Fragment Name R1的表头,对该列按名称进行排序。
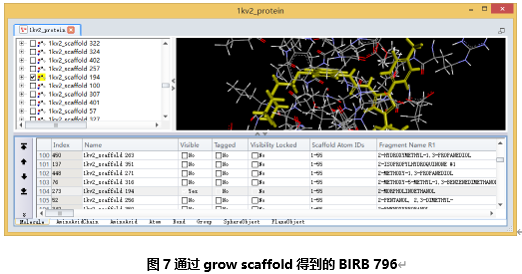
找到2-MORPHOLINOETHANOL并选中该行,点击 (数据窗口左侧)查看分子(图7)。
(数据窗口左侧)查看分子(图7)。
或者采用filter方法。
在表格视图中点击鼠标右键,选择Filter...。
在弹出的窗口中,设置“Fragment Name R1”的Choice为Contains。
设置Value为MORPHO,点击OK。
表格视图会自动按上述设置的限制条件过滤得到符合的分子。
然后选中2-MORPHOLINOETHANOL。
(二)使用自定义反应产生先导化合物
尽管Grow Scaffold的所自带的反应类型已经能够概括药物化学家常用的化学反应,不过你还可以根据自己的需要,轻松地定制自己的反应。接下来我们将介绍如何使用非默认反应类型,来优化优化另一个p38α的抑制剂。
此外,由于p38α激酶linker区上的残基MET109,具有重要作用,我们把它设为必须作用的残基,计算结果中只有与这个残基有相互作用的新分子才会被返回。
1.执行计算
在文件浏览器(File explorer)中找到并双击打开,Samples | Tutorials | Receptor-Ligand Interactions | diverse_sulfides.sd文件。
再切换回刚才的1kv2_complex窗口。
选择菜单栏中的File | Insert From | File...,插入Samples | Tutorials | Receptor-Ligand Interactions | 2yis_scaffold.sd文件。
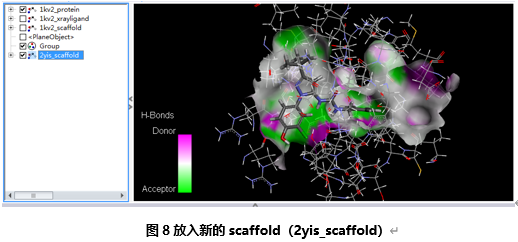
在系统视图中, 勾掉1kv2_scaffold复选框,并选择2yis_scaffold使它显示出来。(图8)
在2yis_scaffold配体中,选中受体表面开口方向上脲基邻位上的笨基上的氢 (H19)。
在工具浏览器(Tool Explorer)中,打开Receptor-Ligand Interactions | Lead Optimization工具栏, 点击Grow Scaffold...打开流程对话框。
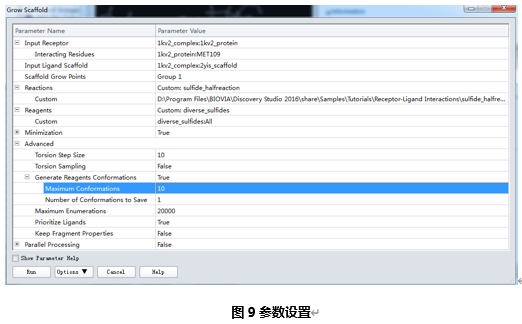
设置Input Receptor参数为1kv2_complex:1kv2_protein。
展开Input Receptor参数组, 将Interacting Residues参数设置为MET109(该group已事先定义好,定义方法:展开1kv2_protein,展开A链,选择Met109残基,在分子窗口中右键|Group,设置group名称为MET109)。
设置Input Ligand Scaffold为1kv2_complex:2yis_scaffold。
设置Scaffold Grow Points,并选择Create New Group From Selection...(刚才选中的H19)。
展开Reactions参数组,先将Custom设置为Samples | Tutorials | Receptor-Ligand Interactions | sulfide_halfreaction.rxn文件,然后再将Reactions参数设置为Custom: sulfide_halfreaction,并勾掉其他参数。(PS:Custom: sulfide_halfreaction只有设置了custom才会出现)
展开Reagent参数组,先将Custom参数设置为diverse_sulfides:All.,然后在Reagents中勾选Custom: diverse_sulfides选项。
将Minimization设为True。
展开Advanced参数组,将Torsion Step Size设为10,Generate Reagents Conformations设为True, 并展开,将Maximum Conformations设为10。
其他参数为默认(图9),点击Run按钮运行作业,点击Background使作业在后台运行。
该作业大概将运行10分钟,最终返回14个结果。
2.查看结果
点击作业浏览器(Job explorer)中刚完成的作业,点击View Results链接。
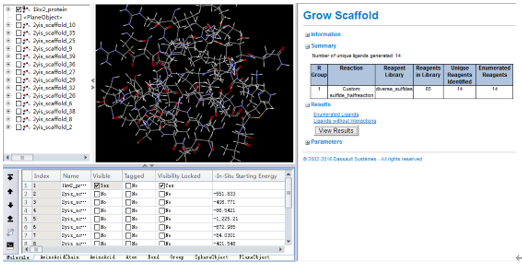
在工具浏览器(Tools Explorers)中,展开Receptor-Ligand Interactions | View Interactions,点击Ligand Interactions。
与前面的步骤一样,点击上述View Interaction工具面板下的 按钮可以观察优化后的分子同蛋白之间的各种非键相互作用。(图10)
按钮可以观察优化后的分子同蛋白之间的各种非键相互作用。(图10)
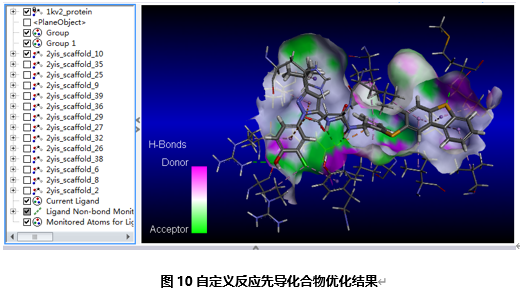
尽管没有化合物与PF-03715455一样,因为diverse_sulfides.sd文件中并没有包含形成PF-03715455所必需的子结构(hydroxyethylsulfanylphenyltriazolopyridinyl)。然而,你会发现一些有趣的新的取代,包括噻唑,benzoxazolinone,吲唑,苯并咪唑,和imidazopyridine子结构。

https://m.sciencenet.cn/blog-3536821-1362300.html
上一篇:Discovery Studio官方教程(Help-Tutorials) 使用LibDock进行快速分子对接(虚拟筛选)
下一篇:Discovery Studio官方教程(Help-Tutorials) 使用CDOCKER进行半柔性分子对接