博文
遗传相互作用(协同致死)在药物组合设计、个体化治疗方面的应用
||||
遗传相互作用(协同致死)可用于开发药物组合、设计靶向治疗、设计个体化治疗策略:
首先,遗传相互作用可以用于药物组合的设计。例如,Whitehurst等人针对药物紫杉醇(paclitaxel),利用协同致死筛选实验筛选得到87个与紫杉醇的作用位点(细胞内微管的动态过程)具有协同致死效应的基因,对这些基因的靶向干预可以将肿瘤细胞对紫杉醇的敏感性提高1000倍以上[1]。这些协同致死基因中含有大量蛋白酶体基因,因此可以据此设计蛋白酶体抑制剂(如bortezomib)与紫杉醇的药物组合,该组合可以充分利用蛋白酶体蛋白质与紫杉醇固有靶点(微管)之间的协同致死效应而发挥作用,其对肿瘤细胞的杀伤效应远远大于单一药物的应用效果,这一推测已得到实验证实[2]。
其次,遗传相互作用可用于通过药物组合增强靶向治疗的效果。例如,厄洛替尼(Erlotinib)是一种小分子口服靶向药物,是首个表皮生长因子(epidermal growth factor receptor,EGFR)酪氨酸激酶的选择性抑制剂。Astsaturov等人利用协同致死筛选实验筛选得到与EGFR具有协同致死效应的蛋白质网络。对这些蛋白质的靶向干预可以显著增强厄洛替尼的肿瘤细胞杀伤效果。针对这些与EGFR具有协同致死效应的基因,其小分子抑制剂(例如,基因STAT3的抑制剂Stattic)与厄洛替尼进行组合,对厄洛替尼的疗效具有显著的增强作用[3]。
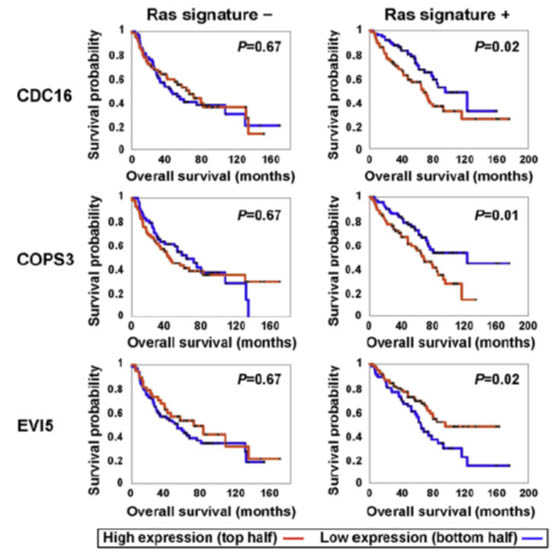
最后,遗传相互作用可用于设计针对性的个体化治疗策略。例如,癌基因Ras的突变是一种在多种类型癌症中广泛存在的现象,Luo等人利用遗传筛选实验,鉴定出一系列与癌基因Ras具有协同致死效应的基因,这些基因主要参与与有丝分裂激酶PLK1以及蛋白酶体相关的生物学过程[4]。图1显示了这些基因(如图中的CDC16/COPS3/EVI5等)与Ras基因的关系[4],依此关系设计个体化治疗策略的基本方法为(以CDC16为例):
step 1, 分析Ras基因未发生突变时,CDC16基因与病人预后之间的相关性。图1所示的图为生存期分析(survival analysis)中常用的K-M图(Kaplan-Meier plot),横坐标表示病人群体的生存时间,纵坐标表示生存概率,图中每个点为病人群体在某一时间段内的存活的比例(概率)。左上角子图显示在Ras基因无突变的病人群体(“Ras signature –”)中CDC16基因表达值与病人生存期之间的关系。CDC16高表达的病人群体的生存曲线用红色连线表示,CDC16低表达的病人群体的生存曲线用蓝色连线表示,图中可以看出这两个群体的生存曲线之间无显著差别(红色连线与蓝色连线交叉在一起)。利用测试生存曲线差异显著性的Log-rank Test进行分析,显示这两个群体生存曲线无显著差别(P-value为0.67),说明在Ras基因未突变的病人群体中,CDC16的基因表达情况与病人的预后无关,推测对Ras基因未突变的病人群体进行针对CDC16的靶向治疗可能导致无效。
step 2,分析Ras基因发生突变时,CDC16基因与病人预后之间的相关性。图1右上角子图显示在Ras基因发生突变的病人群体(“Ras signature +”)中CDC16基因表达值与病人生存期之间的关系。图中可以看出,CDC16高表达的病人(红色连线)与CDC16低表达的病人(蓝色连线)的生存曲线具有显著的差别(红色连线与蓝色连线分离),其Log-rank Test测试P-value为0.02。说明在Ras基因发生突变的病人群体中,CDC16的基因表达情况与病人的预后相关,推测对Ras基因突变的病人群体进行针对CDC16的靶向治疗有效。
step 3, 综合上述分析结果,可以设计如下个体化治疗策略:“在决定进行靶向CDC16的治疗前,进行病人Ras基因突变的检测,如果检测结果呈阳性(Ras发生突变),则采用CDC16靶向治疗;如果检测结果呈阴性(Ras未发生突变),则不考虑采用CDC16靶向治疗(因CDC16靶向治疗对这种病人无效)”。
综上所述,作为一种遗传相互作用,SOD在药物组合、靶向治疗、个体化治疗等方面均具有与协同致死类似的应用潜力。在后续研究中,将结合生物实验研究,对上述基因协同关系的潜在应用进行探索。
图1 利用遗传相互作用(协同致死)设计个体化治疗策略的原理
(引自” A Genome-wide RNAi Screen Identifies Multiple Synthetic Lethal Interactions with the Ras Oncogene”[4])
1. Whitehurst, A.W., et al., Synthetic lethal screen identification of chemosensitizer loci in cancer cells. Nature, 2007. 446(7137): p. 815-9.
2. Davies, A.M., et al., Bortezomib-based combinations in the treatment of non-small-cell lung cancer. Clin Lung Cancer, 2005. 7 Suppl 2: p. S59-63.
3. Astsaturov, I., et al., Synthetic lethal screen of an EGFR-centered network to improve targeted therapies. Sci Signal, 2010. 3(140): p. ra67.
4. Luo, J., et al., A genome-wide RNAi screen identifies multiple synthetic lethal interactions with the Ras oncogene. Cell, 2009. 137(5): p. 835-48.
https://m.sciencenet.cn/blog-508476-582165.html
上一篇:生物信息学 助力 临床数据挖掘
下一篇:microRNAs作为网络化治疗策略的特色
首先,遗传相互作用可以用于药物组合的设计。例如,Whitehurst等人针对药物紫杉醇(paclitaxel),利用协同致死筛选实验筛选得到87个与紫杉醇的作用位点(细胞内微管的动态过程)具有协同致死效应的基因,对这些基因的靶向干预可以将肿瘤细胞对紫杉醇的敏感性提高1000倍以上[1]。这些协同致死基因中含有大量蛋白酶体基因,因此可以据此设计蛋白酶体抑制剂(如bortezomib)与紫杉醇的药物组合,该组合可以充分利用蛋白酶体蛋白质与紫杉醇固有靶点(微管)之间的协同致死效应而发挥作用,其对肿瘤细胞的杀伤效应远远大于单一药物的应用效果,这一推测已得到实验证实[2]。
其次,遗传相互作用可用于通过药物组合增强靶向治疗的效果。例如,厄洛替尼(Erlotinib)是一种小分子口服靶向药物,是首个表皮生长因子(epidermal growth factor receptor,EGFR)酪氨酸激酶的选择性抑制剂。Astsaturov等人利用协同致死筛选实验筛选得到与EGFR具有协同致死效应的蛋白质网络。对这些蛋白质的靶向干预可以显著增强厄洛替尼的肿瘤细胞杀伤效果。针对这些与EGFR具有协同致死效应的基因,其小分子抑制剂(例如,基因STAT3的抑制剂Stattic)与厄洛替尼进行组合,对厄洛替尼的疗效具有显著的增强作用[3]。
最后,遗传相互作用可用于设计针对性的个体化治疗策略。例如,癌基因Ras的突变是一种在多种类型癌症中广泛存在的现象,Luo等人利用遗传筛选实验,鉴定出一系列与癌基因Ras具有协同致死效应的基因,这些基因主要参与与有丝分裂激酶PLK1以及蛋白酶体相关的生物学过程[4]。图1显示了这些基因(如图中的CDC16/COPS3/EVI5等)与Ras基因的关系[4],依此关系设计个体化治疗策略的基本方法为(以CDC16为例):
step 1, 分析Ras基因未发生突变时,CDC16基因与病人预后之间的相关性。图1所示的图为生存期分析(survival analysis)中常用的K-M图(Kaplan-Meier plot),横坐标表示病人群体的生存时间,纵坐标表示生存概率,图中每个点为病人群体在某一时间段内的存活的比例(概率)。左上角子图显示在Ras基因无突变的病人群体(“Ras signature –”)中CDC16基因表达值与病人生存期之间的关系。CDC16高表达的病人群体的生存曲线用红色连线表示,CDC16低表达的病人群体的生存曲线用蓝色连线表示,图中可以看出这两个群体的生存曲线之间无显著差别(红色连线与蓝色连线交叉在一起)。利用测试生存曲线差异显著性的Log-rank Test进行分析,显示这两个群体生存曲线无显著差别(P-value为0.67),说明在Ras基因未突变的病人群体中,CDC16的基因表达情况与病人的预后无关,推测对Ras基因未突变的病人群体进行针对CDC16的靶向治疗可能导致无效。
step 2,分析Ras基因发生突变时,CDC16基因与病人预后之间的相关性。图1右上角子图显示在Ras基因发生突变的病人群体(“Ras signature +”)中CDC16基因表达值与病人生存期之间的关系。图中可以看出,CDC16高表达的病人(红色连线)与CDC16低表达的病人(蓝色连线)的生存曲线具有显著的差别(红色连线与蓝色连线分离),其Log-rank Test测试P-value为0.02。说明在Ras基因发生突变的病人群体中,CDC16的基因表达情况与病人的预后相关,推测对Ras基因突变的病人群体进行针对CDC16的靶向治疗有效。
step 3, 综合上述分析结果,可以设计如下个体化治疗策略:“在决定进行靶向CDC16的治疗前,进行病人Ras基因突变的检测,如果检测结果呈阳性(Ras发生突变),则采用CDC16靶向治疗;如果检测结果呈阴性(Ras未发生突变),则不考虑采用CDC16靶向治疗(因CDC16靶向治疗对这种病人无效)”。
综上所述,作为一种遗传相互作用,SOD在药物组合、靶向治疗、个体化治疗等方面均具有与协同致死类似的应用潜力。在后续研究中,将结合生物实验研究,对上述基因协同关系的潜在应用进行探索。
图1 利用遗传相互作用(协同致死)设计个体化治疗策略的原理
(引自” A Genome-wide RNAi Screen Identifies Multiple Synthetic Lethal Interactions with the Ras Oncogene”[4])
1. Whitehurst, A.W., et al., Synthetic lethal screen identification of chemosensitizer loci in cancer cells. Nature, 2007. 446(7137): p. 815-9.
2. Davies, A.M., et al., Bortezomib-based combinations in the treatment of non-small-cell lung cancer. Clin Lung Cancer, 2005. 7 Suppl 2: p. S59-63.
3. Astsaturov, I., et al., Synthetic lethal screen of an EGFR-centered network to improve targeted therapies. Sci Signal, 2010. 3(140): p. ra67.
4. Luo, J., et al., A genome-wide RNAi screen identifies multiple synthetic lethal interactions with the Ras oncogene. Cell, 2009. 137(5): p. 835-48.
https://m.sciencenet.cn/blog-508476-582165.html
上一篇:生物信息学 助力 临床数据挖掘
下一篇:microRNAs作为网络化治疗策略的特色