博文
新型DGE产品与与传统21bp DGE间的差别
|||
随着高通量测序的普及,数字基因表达谱(DGE)也走进了更多的实验室,用以代替传统的定量PCR。但目前测序市场上DGE的概念十分模糊不清,价格也参差不齐,导致很多研究者不知该如何选择,这里通过查阅一些文献,大概解释一下现在市面上新型的DGE产品与传统21bp DGE之间的差别,以供各位研究者参考。
数字基因表达谱(Digital Gene Expression Profiling,DGE),是基于高通量测序技术,研究某物种特定组织在特定状态下的基因表达情况。2009年Morrissy等[1]在GenomeResearch上发表了使用NlaIII和MmeI双酶切的方法构建插入片段21bp的tag文库,被称做“传统21bp的DGE”。目前,随着建库测序成本的降低,DGE建库方便也从传统的21bp文库转向均一性更好的普通转录组文库。其优势比较见下:
传统的21bp DGE文库:
建库流程:传统的21bp DGE,建库基于酶切位点CATG,建库的时候先将mRNA反转为cDNA,再使用NlaIII和MmeI进行双酶切,构建插入片段21bp的文库,然后使用SE50 或 SE36测序。
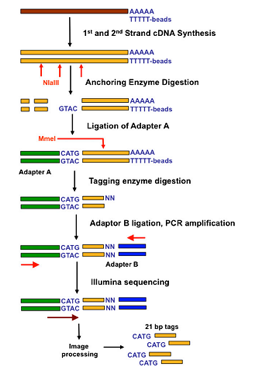
图1 21bp的tag-seq文库构建流程[1]
缺点:
1. mRNA先反转为cDNA,再酶切为短片段。由于mRNA的片段较长,并且存在二级结构,这种方法会影响反转录酶的活性,影响反转效率[2]。
2. 酶切建库,基于的是cDNA上四碱基CATG的分布,这个分布情况是否均一,直接影响了建库的片段均一性。比如有些区域没有该酶切位点,那么建库的时候就不能被捕获,信息有所丢失。另外,这种建库方式其实最初是在人这一物种上验证,所以对于其他的物种,均一性情况并没有进行过评估[1]。
3. 测序:如果构建21bp的tag文库,测序使用SE36 或者SE50,那么会有十多bp的碱基是adapter序列,这部分数据没有意义。
最新构建方法:(与构建转录组文库方法一致)
建库流程:纯化得到的mRNA,均一性的打断为200bp的片段,再进行cDNA合成,构建转录组文库。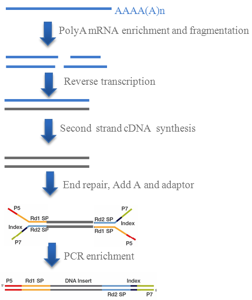
图2 随即打断的普通转录组构建流程
优势:
1. 纯化得到的mRNA,先进行打断,消除mRNA的二级结构,且短片段时,提高反转效率。
2. 文库构建针对全部的mRNA,不依赖于特定部位的酶切位点,提高建库均一性,没有CATG酶切位点的地方也能被测到。插入片段为200bp。
3. 测序:采用SE100/SE50测序,reads较长,对基因组mapping的时候更加准确。且全部碱基都可用于后续分析。
参考文献:
[1]AS Morrissy, RD Morin, A Delaney, et al. Lu X,et al. Next-generation tagsequencing for cancer gene expression profiling. Genome Research, 2009, 19:1825-1835.
[2]Z Wang, M Gerstein and M Snyder. RNA-Seq: a revolutionary tool for transcriptomics.Nature Reviews, 2009, 10:57-63.
https://m.sciencenet.cn/blog-1020398-716953.html
下一篇:选择生物信息培训班注意事项