博文
GWAS分析中:为何有些SNP效应值大却不显著?
||
之前的GWAS分析中,我查看结果时有过这个疑问,随着不断的学习理解,我已经知道了为什么,这里将我思考的结果分享一下。
1. 错误的理解
某一个SNP,效应值(Effect)越大,就越显著???
「事实上:」效应值和显著性是两码事!
2. 用代码说话
GWAS分析中,最简单的就是一般线性模型(GLM),而GLM模型进行的GWAS分析中,用R语言实现的代码如下:
mod_M7 = lm(phe.V3 ~ M7_1,data=dd)
summary(mod_M7)
「结果:」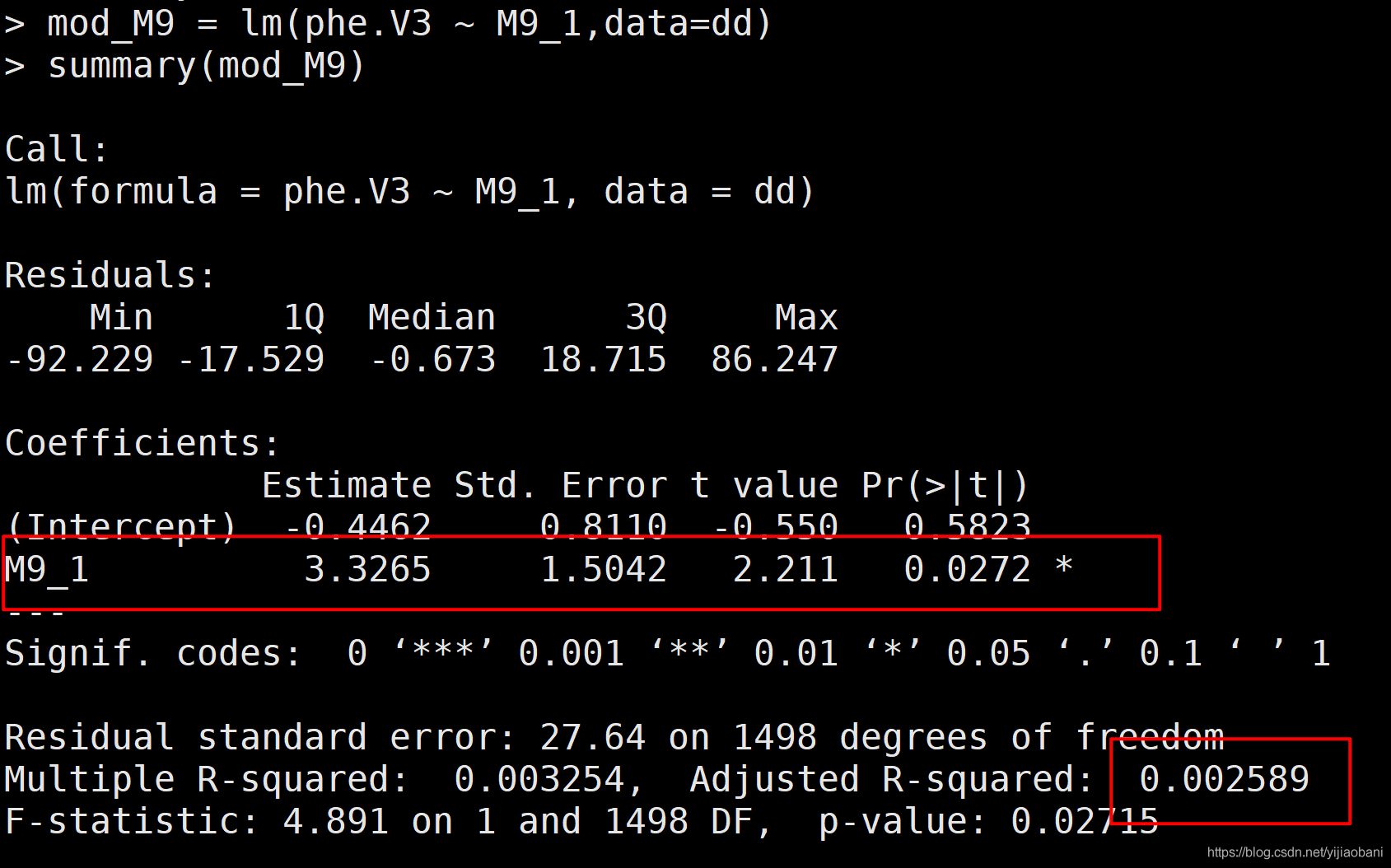 这个里面:
这个里面:
Estimate就是效应值:3.3265,这个就是SNP M9的效应值 Pr就是P值:0.0272,这个就是SNP M9的P值
可以看到,这两个是两个指标,他们之间没有必然的联系。
3. GWAS分析的思路
1,将SNP的分型转化为 0, 1, 2的形式,主效纯合(major)编码为0,杂合编码为1,次等位纯合编码为22,x变量为 0,1,2的数值,y变量为性状表型值3,对x和y做回归分析 y ~ a*x + b,a为效应值,a是否显著的P值,为SNP的P值
4. SNP效应值很大,却不显著
# SNP 分型
set.seed(666)
x = rep(c(0,1,2),3)
x
# 模拟表型值
y = 12*x + rnorm(9)*10
# 作回归分析
mod = lm(y ~ x)
summary(mod)
# 作图
dd = data.frame(x,y)
dd
ggplot(dd,aes(x=x,y=y)) + geom_point() + stat_smooth(method='lm',formula = y~x,colour='red')
看一下这个数据:
> dd
x y
1 0 7.533110
2 1 32.143547
3 2 20.448655
4 0 20.281678
5 1 -10.168745
6 2 31.583962
7 0 -13.061853
8 1 3.974804
9 2 6.077592
计算每种分型对应的表型平均值:
> # 平均值
> aggregate(y~x,data = dd,mean)
x y
1 0 4.917645
2 1 8.649869
3 2 19.370070
可以看出,分型0对应的是4.9,最小,分型1对应的是8.6,分型2对应的是19.3,趋势是比较明显的。
「看一下回归分析的结果:」
> # 作回归分析
> mod = lm(y ~ x)
> summary(mod)
Call:
lm(formula = y ~ x)
Residuals:
Min 1Q Median 3Q Max
-21.148 -12.128 2.243 13.379 21.164
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.753 8.558 0.439 0.674
x 7.226 6.629 1.090 0.312
Residual standard error: 16.24 on 7 degrees of freedom
Multiple R-squared: 0.1451, Adjusted R-squared: 0.023
F-statistic: 1.188 on 1 and 7 DF, p-value: 0.3118
可以看出,效应值为7.226,但是P值却是0.312,不显著。
所以说,效应值大的SNP位点,不一定是显著的。
「为何会出现这种情况呢?」我们画一个散点图看一下分布情况:
library(ggplot2)
ggplot(dd,aes(x=x,y=y)) + geom_point() + stat_smooth(method='lm',formula = y~x,colour='red')
 可以看到,分型为1的表型值,有一个非常高,达到了31,相当于在进行
可以看到,分型为1的表型值,有一个非常高,达到了31,相当于在进行T检验时,标准误se比较高,导致P值较大,不显著。
5. SNP效应值很小,却极显著
我们还模拟数据:
# SNP 分型
set.seed(666)
x = rep(c(0,1,2),3)
x
# 模拟表型值
y = 0.1*x + rnorm(9)*0.01
# 作回归分析
mod = lm(y ~ x)
summary(mod)
# 作图
dd = data.frame(x,y)
dd
library(ggplot2)
ggplot(dd,aes(x=x,y=y)) + geom_point() + stat_smooth(method='lm',formula = y~x,colour='red')
# 平均值
aggregate(y~x,data = dd,mean)
「模拟的数据如下:」
> dd
x y
1 0 0.00753311
2 1 0.12014355
3 2 0.19644866
4 0 0.02028168
5 1 0.07783126
6 2 0.20758396
7 0 -0.01306185
8 1 0.09197480
9 2 0.18207759
计算每种分型对应的表型平均值:
> aggregate(y~x,data = dd,mean)
x y
1 0 0.004917645
2 1 0.096649869
3 2 0.195370070
可以看到,三种基因型,阶梯感也很强。
「回归分析结果:」
> # 作回归分析
> mod = lm(y ~ x)
> summary(mod)
Call:
lm(formula = y ~ x)
Residuals:
Min 1Q Median 3Q Max
-0.021148 -0.012128 0.002243 0.013379 0.021164
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 0.003753 0.008558 0.439 0.674
x 0.095226 0.006629 14.365 1.89e-06 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 0.01624 on 7 degrees of freedom
Multiple R-squared: 0.9672, Adjusted R-squared: 0.9625
F-statistic: 206.4 on 1 and 7 DF, p-value: 1.886e-06
可以看出: SNP的效应值:0.095 SNP的P值:1.89e-6(达到极显著水平)
「作图看一下:」 虽然回归系数(SNP效应值很小),但是每个分型对应的表型值相差不多,组内变异(同一种基因型的表型值)远远小于组间变异(不同SNP分型对应的表型值),导致进行
虽然回归系数(SNP效应值很小),但是每个分型对应的表型值相差不多,组内变异(同一种基因型的表型值)远远小于组间变异(不同SNP分型对应的表型值),导致进行T检验时,达到极显著水平。
这也就出现了SNP的效应值很低,但是却达到了极显著水平。
6. 从生物学水平如何解释?
可以这样理解,「SNP效应值小但显著的位点:」属于对表型值是有直接影响的,但是影响的效应较小,但是比较稳定,所以才表现出极显著,而效应小
「SNP效应大但不显著的位点:」有可能SNP是有大效应的,但是容易受到环境影响,或者由于群体的结构(某个群体内聚集),或者由于某些异常值导致这种情况。也有可能本身就是不显著的。
「林子大了,什么鸟都有!」毕竟SNP都是上万,上十万,上百万的量。而表型值都是上百,上千,很少达到上万的,这就存在很多种情况,导致有些SNP效应值很大,但是不显著,有些SNP效应值很小,但是极显著。这里需要区分对待一下。
如果有很多SNP出现这种情况,必然有妖,查看是否过滤了maf,是否对基因型进行了填充,是否表型数据有异常,如果都检查过了,出现这种情况,我们也只能说:it happens,有时候会出现,这不是不可解释的现象,否者我写这篇文章的意义在哪里呢?除了灌水,还有炫技啊
7. GWAS系列相关
笔记 | GWAS 操作流程4-2:LM模型linear+数值协变量
笔记 | GWAS 操作流程4-4:LM模型+数值+因子协变量
笔记 | GWAS 操作流程4-5:LM模型+数值+因子+PCA协变量
https://m.sciencenet.cn/blog-2577109-1250860.html
上一篇:R语言计算玉米测交试验的配合力
下一篇:R语言tryCatch使用方法:判断Warning和Error