博文
关于16s扩增区域选择的文章
||||
文章:Primer and platform effects on 16SrRNA tag sequencing
杂志:Frontiers in Microbiology 2015
研究方案:three different amplification primer sets (targeting V4, V6–V8, and V7–V8)三个区域 、two sequencing technologies (454 pyrosequencing and Illumina MiSeq) 454和Miseq测序、DNA from a mock community containing a known number of species as well as complex environmental samples(mock community).
研究结论:1)测序错误的比较
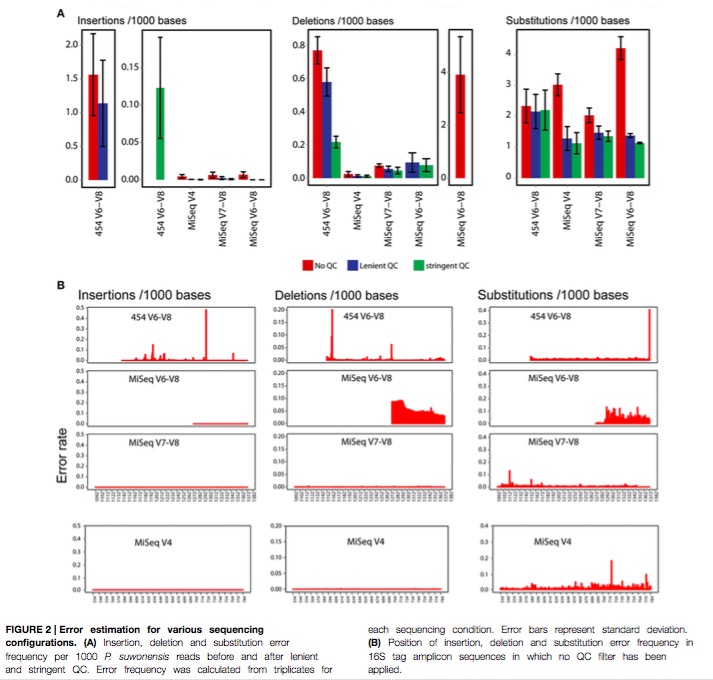
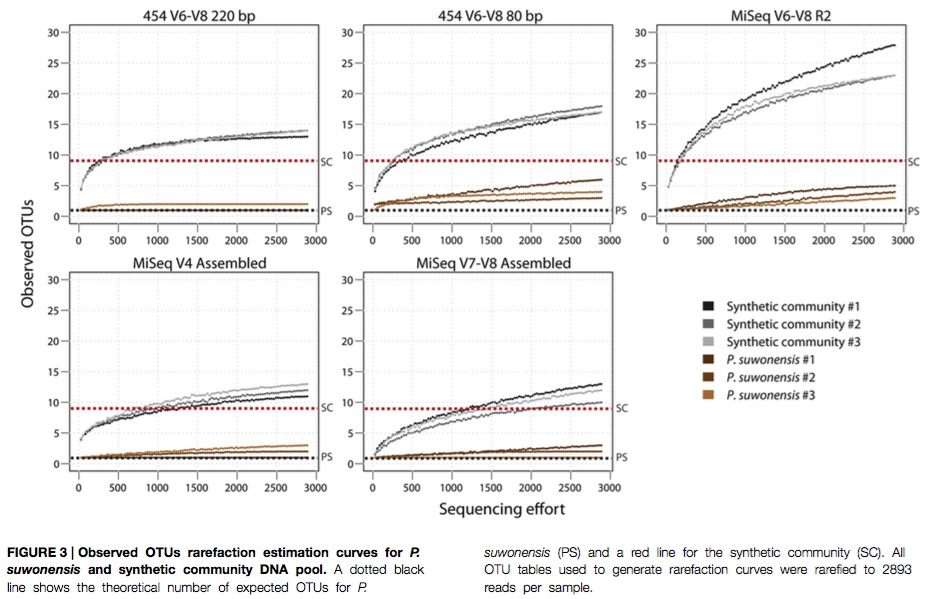
2)稀释曲线的比较
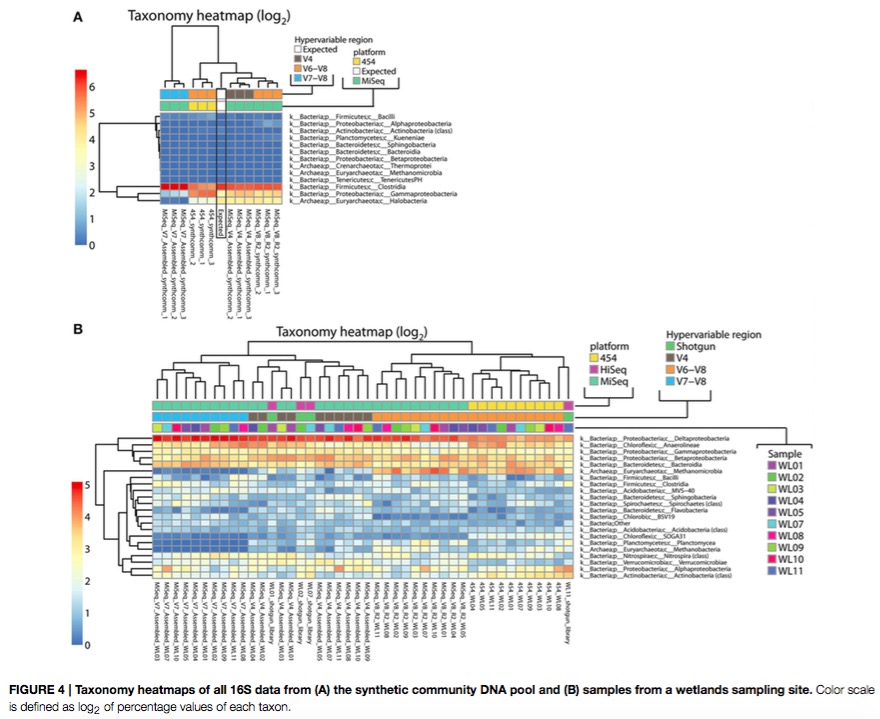
3)物种分类结果比较,不同的区域物种分类有较大区别
文章:Comparison of two next-generation sequencingtechnologies for resolving highly complexmicrobiota composition using tandem variable 16S rRNA gene regions
杂志:Nucleic Acids Research 2010
研究结论:In silico evaluations predicted that the V3/V4 and V4/V5 regions would provide the highest classi-fication accuracies for both technologies. 计算的策略发现V3/V4、V4/V5的效果最好。但在实验中V3/V4会带来较大的bias,However, experimental sequencing of the V3/V4 region revealed significant amplification bias compared to the other regions, emphasising the necessity forexperimental validation of primer pairs.
具体结果:1)reads稍长,分类效果较好
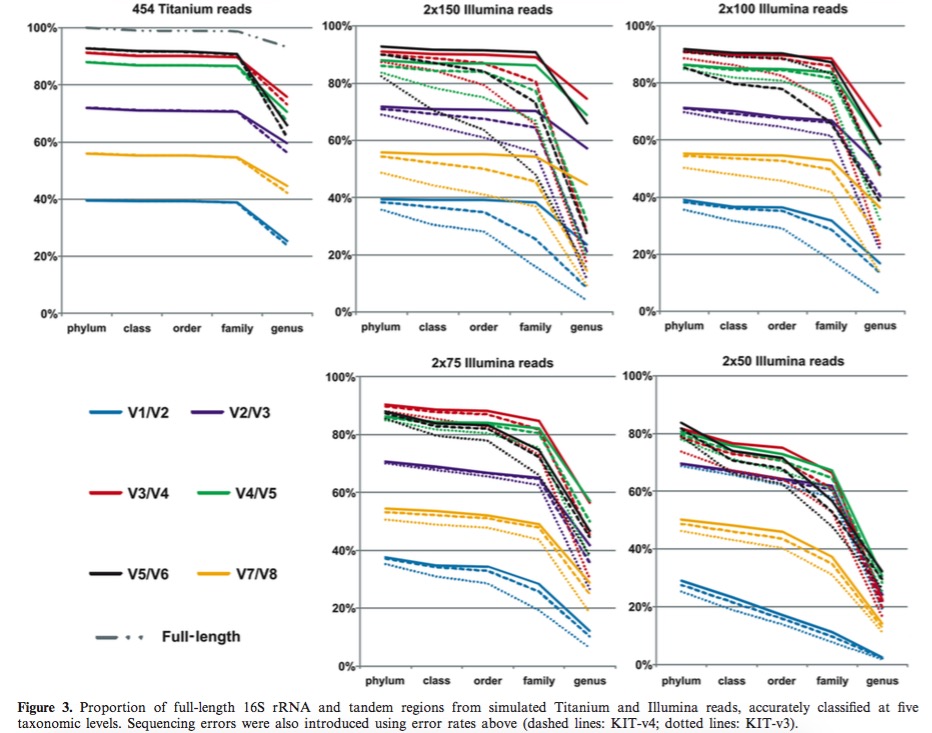
2)454的数据要好于illumina的数据
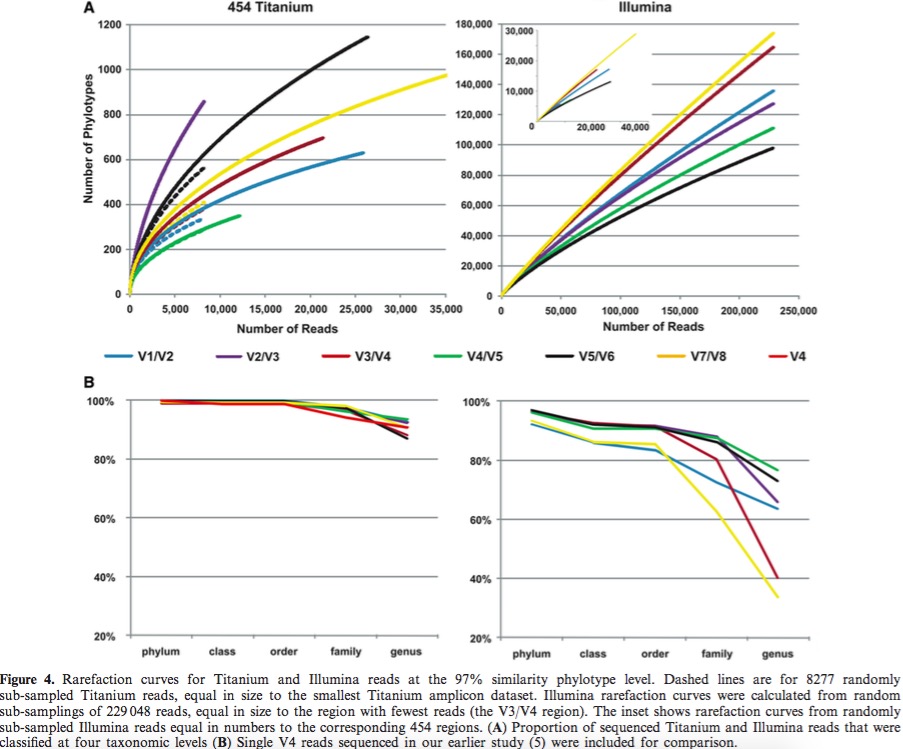
3)不同区域物种分类效果存在较大差异
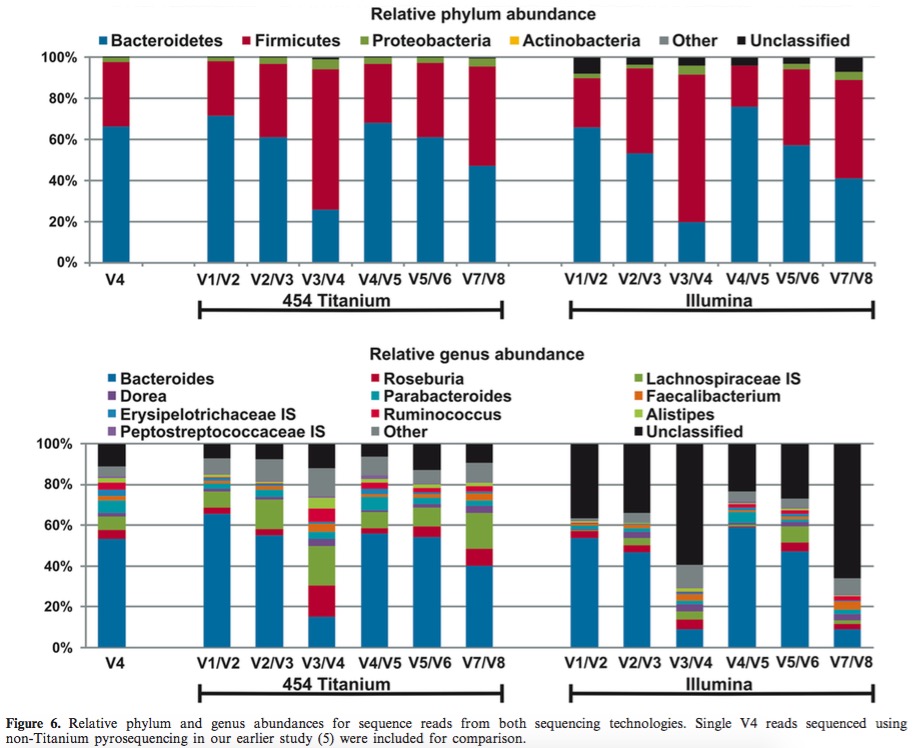
文章:Evaluation of the RDP Classifier Accuracy Using 16S rRNA GeneVariable Regions
杂志:Metagenomics 2012
利用SILVA的数据库数据,以E. Coli的16s序列为基础作为位置坐标去界定V1、V2...V9各个区域,用RDP进行分类,最终得到的结果如下,组合区域中V3-V5的效果较好,单个区域中V2、V4的效果较好。
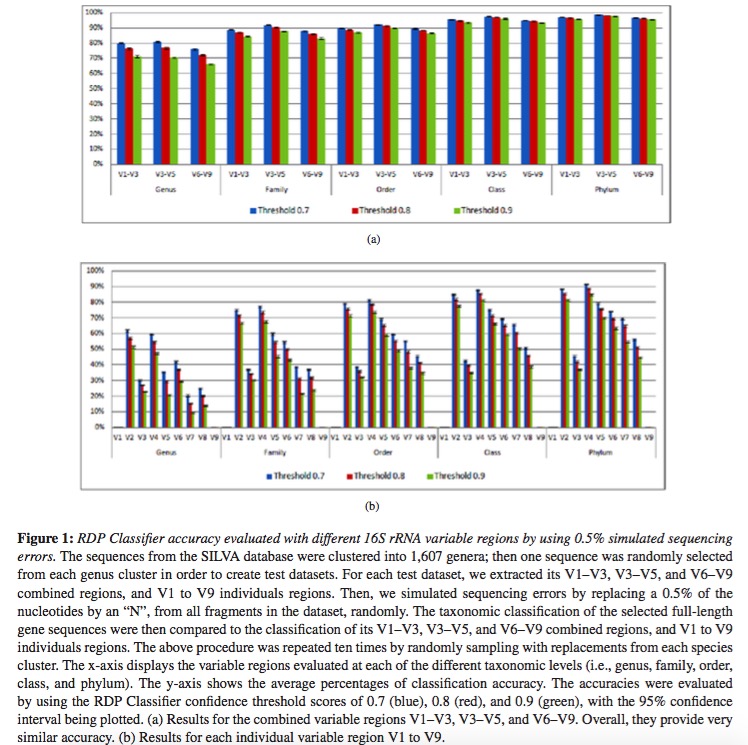
此处的研究未考虑样品实际处理过程中的引物扩增效率情况、不同分类工具的效果;
https://m.sciencenet.cn/blog-306699-989048.html
上一篇:微生物数据的可视化工具
下一篇:pandaseq在linux环境下的安装