博文
Molecular Plant:全基因组复制事件之后基因对间的功能分化
|||
Functional Divergence Between Subgenomes and Gene Pairs After Whole Genome Duplications
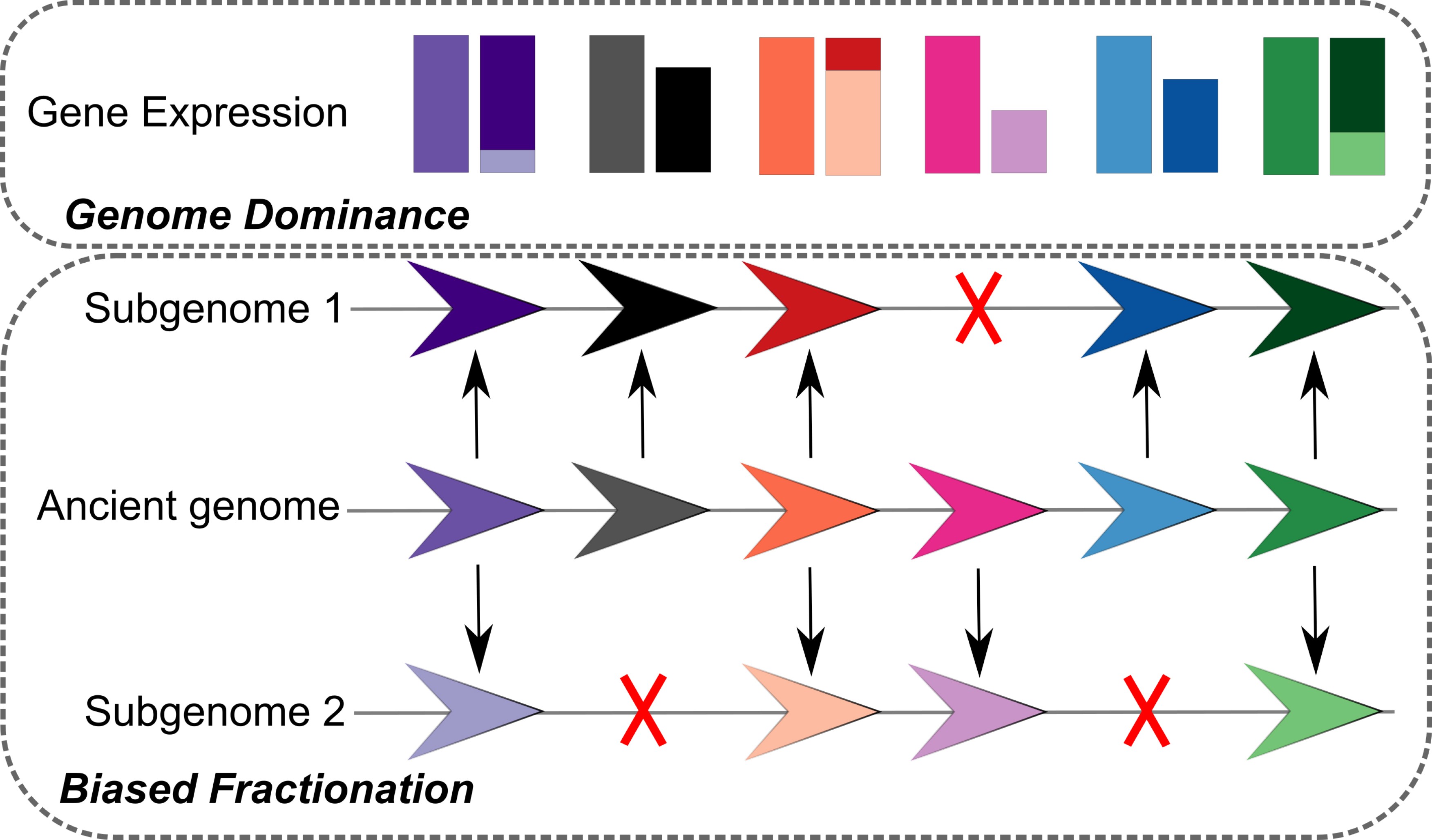
Gene loss following whole genome duplication (WGD) is often biased, with one subgenome retaining more ancestral genes and the other sustaining more gene deletions. While bias toward the greater expression of gene copies on one subgenome can explain bias in gene loss, this shifts the question to what drives differences in gene expression levels between subgenomes. Differences in chromatin markers (染色质标记) and epigenetic signals (表观遗传信号) between subgenomes in several model species are now being identified that may provide an explanation for bias in expression between subgenomes. WGDs can be classified into duplications with higher, biased gene loss and bias in gene expression between subgenomes versus those with lower, unbiased rates of gene loss and an absence of detectable bias between subgenomes; however, the originally proposed link between these two classes and whether WGD results from an allo- or autopolyploid event (异源、同源多倍体事件) is inconsistent with recent data from the allopolyploid Capsella bursa-pastoris (荠菜). The Gene Balance Hypothesis (基因平衡理论) can explain bias in the functional categories of genes retained following whole genome duplication, the difference in gene loss rates between unbiased and biased whole genome duplications, and how plant genomes have avoided being overrun (泛滥) with genes encoding dose-sensitive (剂量敏感) subunits of multiprotein complexes. Comparisons of gene expression patterns between retained transcription factor pairs in maize suggest the high degree of retention for WGD-derived pairs of transcription factors may instead be explained by the older duplication-degeneration-complementation model.
全基因组复制事件之后的基因丢失通常是具有偏性的,其中一个亚基因组保留了更多的祖先基因,而另外一个亚基因组上则有更多的基因删除。亚基因组上对于基因拷贝更高的表达偏性可以解释基因丢失的偏性,这就产生了一个问题:什么驱动了不同亚基因组上基因表达水平的差异?最近在一些模式物种中鉴定到的亚基因组间染色质标记和表观遗传信号差异可能为解释不通亚基因组间基因表达偏性提供新的见解。全基因组复制事件可以划分为两类,一类是亚基因组间具有更高、有偏性的基因丢失和基因表达偏性;另一类是更低、无偏性的基因丢失和亚基因组间检测不到偏性。然而,原本提出了这两类之间的的联系以及异源、同源多倍体事件所产生的全基因组复制都与近期从异源多倍体植物荠菜中获得的一些数据不一致。基因平衡理论可以解释全基因组复制事件之后保留基因的功能偏性,以及无偏性和有偏性全基因组复制之间基因丢失速率的差异,还有植物基因组为何能够避免编码多蛋白复合物剂量敏感亚基基因的使用泛滥。玉米中保留的转录因子基因对表达模式的对比研究显示全基因组复制导致的转录因子基因对高程度的保留可能用老的重复-退化-互补模型来解释更为合理。
基因平衡理论(Gene Balance Hypothesis):在全基因组复制事件之后,基因的保留是不均衡的,一些参与编码大分子复合物中的蛋白亚基或者信号转导途径,转录调控网络中的基因会被保留下来以维持这些生命活动的稳定进行。
重复-退化-互补模型(duplication-degeneration-complementation model):旁系同源基因发生了亚功能化之后而分担了祖先基因功能,并且以功能互补的方式完成祖先基因功能。
个人简介:2008年,康奈尔大学,学士,方向:植物生物、遗传及发育;2012年,加利福尼亚大学伯克利分校,博士,方向:植物生物学;2013年,美国国家科学院植物基因组博士后研究员。
doi: http://dx.doi.org/10.1016/j.molp.2017.12.010
https://m.sciencenet.cn/blog-3158122-1091333.html
上一篇:Plant Biotechnol J:lncRNA在棉花抗真菌病害中的作用
下一篇:New Phytologist:黄瓜CsLFY基因作用于茎尖分生维持
全部作者的其他最新博文
- • Plant Physiology:CsMADS3促进柑果中的叶绿素降解和类胡萝卜素合成(华中农业大学)
- • Molecular Plant:LBD11-ROS反馈调节作用于拟南芥的维管形成层增殖和次生生长(浦项科技大学)
- • Science Advances:根结线虫通过调控植物的CLE3-CLV1模块,促进侵染进程(日本熊本大学)
- • The Plant Cell:拟南芥P小体组分通过对FLC的转录调控,影响开花时间(安徽农业大学)
- • Nature Communications:油菜素内酯参与植物营养生长期转变的分子机制解析(浙江农林大学)
- • Current Biology:光合作用产生的蔗糖驱动侧根“生物钟”(德国弗莱堡大学)