博文
可爱龙实验室再发《科学》论文,解析Craspase系统机制
 精选
精选
||
众所周知,CRISPR-Cas技术早已成为生命科学领域应用最广泛的技术之一,目前基于CRISPR-Cas的基因编辑技术已广泛应用于基因校正,遗传育种等各领域。
目前大家所使用的CRISPR-Cas技术都是基于CRISPR系统的精准靶向和sgRNA介导的核酸酶酶展开的。如果能找到一种sgRNA介导的蛋白酶,那将是生物医疗史上新的一页。
北京时间2022年8月26日凌晨2时,美国康奈尔大学可爱龙实验室联合荷兰代尔夫特理工大学Stan J.J Brouns实验室,在Science杂志以长文形式发表了题为“Craspase is a CRISPR RNA-guided, RNA activated protease”的研究论文。 该论文报道了Craspase(CRISPR偶联的蛋白酶新系统,CRISPR associated Caspase)是一个gRNA引导靶向,并且受到靶向RNA激活的蛋白酶,该蛋白酶受到激活之后可以对天然的蛋白底物进行切割并诱导细胞死亡。在精准医疗新时代背景下,该工具有望引领出全新的精准医疗思路。

最近几年,得益于基因库的爆炸性增长,越来越多的CRISPR-Cas相关的新系统被发现【1】。比如最近两年检索出一种全新的系统type III-E。该系统拥有四个Cas7、一个Cas11以及一个功能位置的insertion结构域。然而在该系统中,这些原本属于独立亚基的组分全部串联成为一个氨基酸长链,成为单亚基模式。
该系统的CRISPR相关蛋白被命名为Cas7-11, 也有一个好听的名字:gRAMP (“重复序列相关的巨大的神奇蛋白”, Giant Repeat Associated Mysterious Protein)。该蛋白被证实拥有RNA靶向能力,并在真核细胞内已展现出应用价值【2-4】。这个系统最大的亮点,是其CRISPR-Cas系统附近的蛋白酶TPR-CHAT,前期的研究表明,这个蛋白酶可以与CRISPR系统形成一个复合物【4】。然而之前还没有找到该系统的蛋白酶活性,或者说还不清楚是靠什么机制去激活该蛋白酶。
美国康奈尔大学可爱龙教授团队胡纯一博士借助冷冻电镜,首先解析了巨大神奇蛋白gRAMP的结构,可以区分出四个序列完全不同的Cas7.1, Cas7.2,Cas7.3, Cas7.4,以及一个Cas11和一个insertion结构域。比较有意思的是,这个系统的guide RNA 5’端具有比其他type III长的多的handle,长度达到了14~20个碱基,而其他Type III类型只有8个碱基【5】。此外,该系统guide RNA的handle区域,前两位(-1和-2)碱基是暴露出来的,可以用于碱基配对,该部分序列被成为PFS区域(protospacer flanking sequence)。见图一。
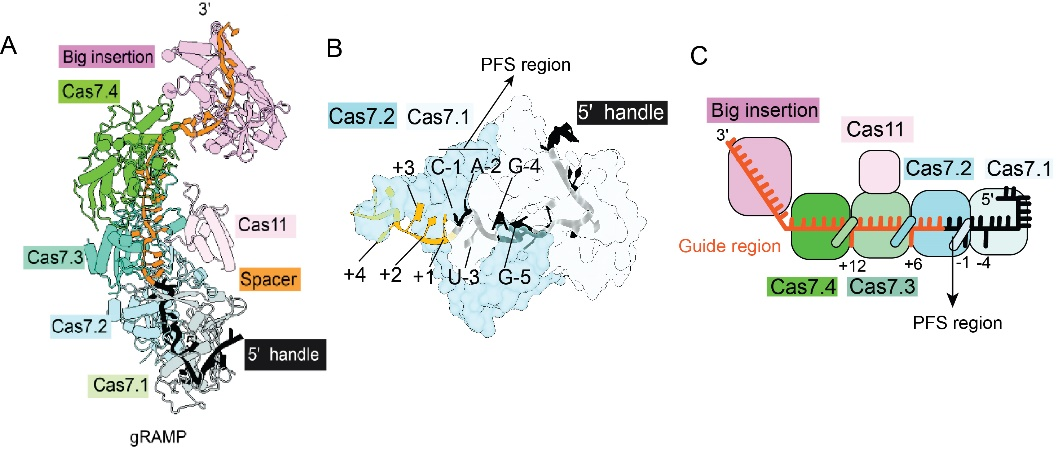
Δ 图一。巨大神奇蛋白gRAMP结构示意图。A.gRAMP蛋白结构全景图。B. guide RNA 5’handle结构特写。PFS区-1,-2是暴露的可以用于RNA靶向。C. gRAMP结构组织示意图。
后续,胡纯一博士等人,继续对Craspase进行了全方位的解析,通过结构比对,发现Craspase只有在结合non-matching PFS的RNA底物时才会发生巨大的构象变化,该类RNA底物可以通过non-matching的PFS的介导靶向RNA的3’端与开关螺旋switch heliex发生空间冲突,这会刺激这个开关螺旋进行重排和位移,而这种构象改变会以一种接力的方式最后引起蛋白酶活性中心的激活(图二)。
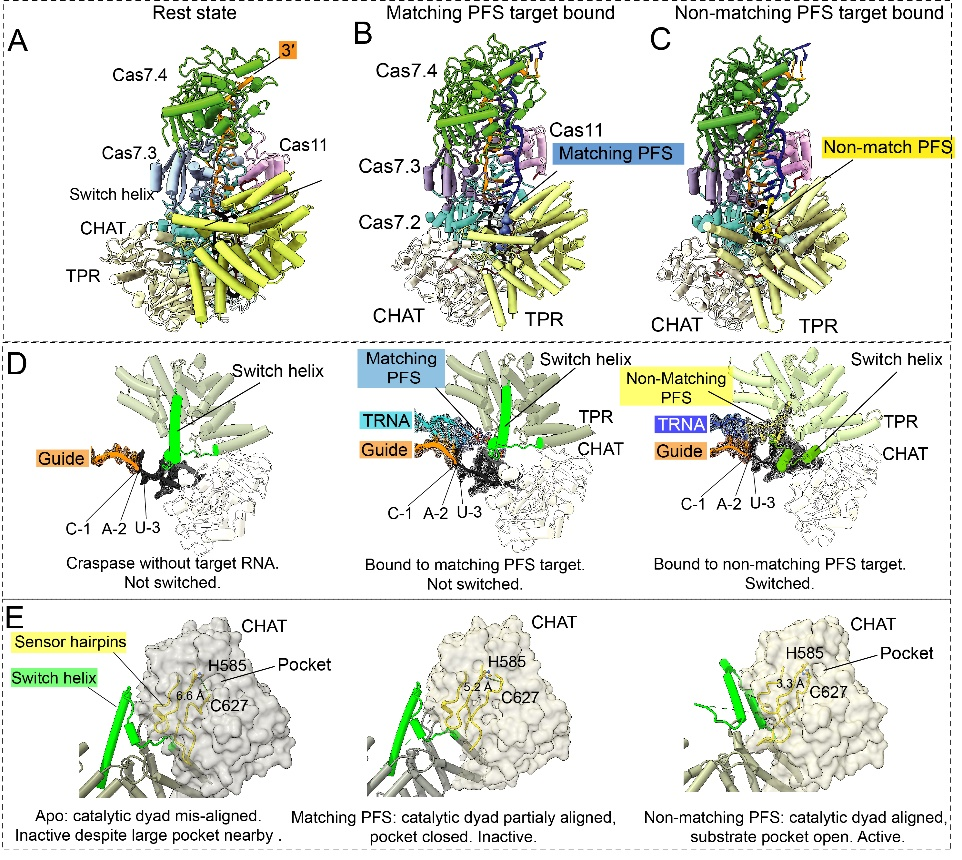
Δ 图二。Craspase蛋白酶受到RNA靶向激活机制。A.静息状态Craspase结构图。B.Matching PFS target RNA结合状态Craspase结构图。C. Non-matching PFS target RNA结合状态Craspase结构图。D. 不同状态下开关螺旋构象图。E.不同状态下蛋白酶催化中心结构图。
通过原子分辨率的结构比较,团队发现和静息状态比,靶向“non-matching”非我序列RNA之后,蛋白酶活性催化二联体组氨酸-半胱氨酸的距离从6.6埃缩小到3.3埃,从而可以实现对蛋白多肽链的攻击。
最后一个关键问题,这个蛋白酶的底物是什么?团队通过体内体外筛选,成功筛选到该Craspase系统蛋白酶底物,名为Csx30。该Craspase系统唯有在结合了“非我”RNA之后能快速特异性地对该蛋白底物进行切割。因此这个结果从生化水平非常确切地验证了该蛋白酶系统受到CRISPR系统的精准调控,最后成为靶向RNA激活的蛋白酶。此外,该团队还从结构出发,设计出了一些多肽序列,在体外也能在靶向RNA激活的情况下实现蛋白酶的精准切割。这在一定程度上暗示了该系统非常有潜力成为蛋白定向降解的新工具(图三),前景十分广大。
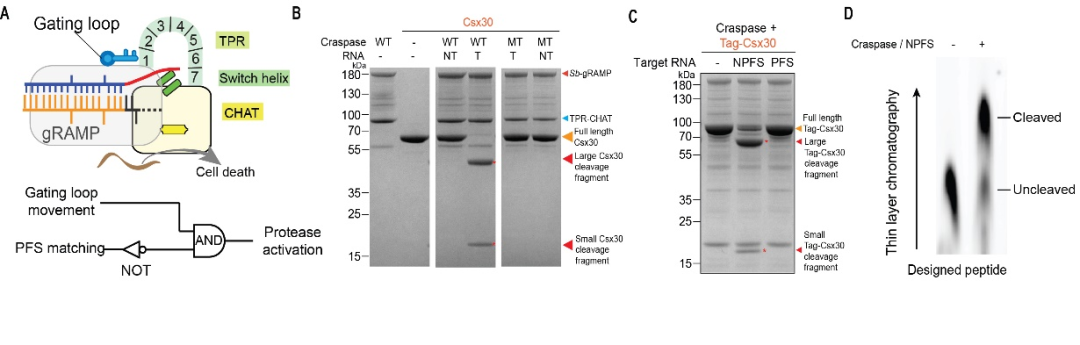
Δ 图三。Craspase蛋白酶激活并且切割蛋白底物。A. Craspase系统蛋白酶激活模式。需要同时至少满足RNA靶向以及PFS区前两位碱基错配。B. Craspase系统切割Csx30需要RNA激活。C. Craspase系统切割Csx30严格通过PFS区的检测来区分自我和非我序列,避免脱靶切割。D.设计工程化多肽可被Craspase系统靶向切割。
CRISPR-Cas系统首先使用Cas1-Cas2从噬菌体内摄取DNA片段整合进入自己的CRISPR区,形成spacer【6】。CRISPR区转录成为crRNA被加工成为guide RNA。Cas蛋白利用guide RNA对噬菌体进行精准靶向和消灭【7】。简言之,CRISPR-Cas系统的初衷实际上就是原核生物一种获得性免疫系统,最终都是用来对抗外来侵略的。因此最后一个终极问题,这个蛋白酶与免疫功能是否相关?
关于这个问题,在8月18日和8月22日,bioRxiv预印本网站上,分别上传的日本东京大学Hiroshi Nishimasu、美国麻省理工大学 Jonathan S. Gootenberg团队合作题为“RNA-triggered protein cleavage and cell death by the RNA-guided type III-E CRISPR-Cas nuclease-protease complex”,以及北京化工大学冯越教授与清华大学杨茂军教授合作题为“Structural and functional insights into the type III-E CRISPR-Cas immunity”的两篇重量级论文(如下图),分别在Desulfonema ishimotonii和Candidatus “Scalindua brodae”(与本篇Science论文使用了同一物种)物种中都观察到了一致的现象。在PFS non-matching的RNA靶向之后,都会激活Caspase-like蛋白酶并切割蛋白底物Csx30。并且这两篇论文都同时明确证实了Csx30在受到Craspase蛋白酶切割之后会诱导细胞死亡。因此这确切的证明这个蛋白酶的确是用来防御噬菌体师兄abortive infection。

总之,这篇Science论文,全面阐述了Craspase系统是如何通过guide RNA引导,并且受到靶向RNA激活的蛋白酶活性的详细机制,该蛋白激活之后会切割蛋白底物,诱导细胞死亡,因此该项工具在未来有可能作为新的精准医疗工具。此外,若在蛋白酶特异性上进化出更加多样性,有望翻开蛋白定向降解的新篇章。

类比图:巨大神秘蛋白gRAMP可以靶向和消除噬菌体RNA。巨人还携带了另一利器:TPR-CHAT蛋白酶。该蛋白酶可以被靶向RNA激活并实现对蛋白底物的切割。图源《巨人战纪》,可爱龙实验室提供。
相关论文信息:
Doi:10.1126/science.add5064
参考文献
1. Makarova, K.S., Wolf, Y.I., Iranzo, J., Shmakov, S.A., Alkhnbashi, O.S., Brouns, S.J.J., Charpentier, E., Cheng, D., Haft, D.H., Horvath, P. et al. (2020) Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants. Nature reviews. Microbiology, 18, 67-83. 2. Kato, K., Zhou, W., Okazaki, S., Isayama, Y., Nishizawa, T., Gootenberg, J.S., Abudayyeh, O.O. and Nishimasu, H. (2022) Structure and engineering of the type III-E CRISPR-Cas7-11 effector complex. Cell, 185, 2324-2337 e2316. 3. Ozcan, A., Krajeski, R., Ioannidi, E., Lee, B., Gardner, A., Makarova, K.S., Koonin, E.V., Abudayyeh, O.O. and Gootenberg, J.S. (2021) Programmable RNA targeting with the single-protein CRISPR effector Cas7-11. Nature, 597, 720-725. 4. van Beljouw, S.P.B., Haagsma, A.C., Rodriguez-Molina, A., van den Berg, D.F., Vink, J.N.A. and Brouns, S.J.J. (2021) The gRAMP CRISPR-Cas effector is an RNA endonuclease complexed with a caspase-like peptidase. Science, 373, 1349-1353. 5. You, L., Ma, J., Wang, J., Artamonova, D., Wang, M., Liu, L., Xiang, H., Severinov, K., Zhang, X. and Wang, Y. (2019) Structure Studies of the CRISPR-Csm Complex Reveal Mechanism of Co-transcriptional Interference. Cell, 176, 239-253 e216. 6. Hu, C., Almendros, C., Nam, K.H., Costa, A.R., Vink, J.N.A., Haagsma, A.C., Bagde, S.R., Brouns, S.J.J. and Ke, A. (2021) Mechanism for Cas4-assisted directional spacer acquisition in CRISPR-Cas. Nature, 598, 515-520. 7. Hu, C., Ni, D., Nam, K.H., Majumdar, S., McLean, J., Stahlberg, H., Terns, M.P. and Ke, A. (2022) Allosteric control of type I-A CRISPR-Cas3 complexes and establishment as effective nucleic acid detection and human genome editing tools. Molecular cell, 82, 2754-2768 e2755.
https://m.sciencenet.cn/blog-3423233-1352690.html
上一篇:《细胞报告》:陈文标团队揭示巨噬细胞和中性粒细胞参与营养过剩诱导胰岛β细胞死亡过程的新机制
下一篇:《细胞报告》:孙中生团队揭示bHLH转录因子NPAS3在星形胶质细胞发生及自闭症中的作用机制