博文
DNA序列比对可能出现的问题
||||
DNA序列比对可能出现的问题
如果比对出来的序列,保守位点较少(在mega软件中查看,序列顶端的*较少,如下图所示)

说明序列存在一定的问题,需要进一步核对序列,找到原因后方能进行后续操作。
第一种原因,可能存在重复序列,一些较长的序列不同的区域可能和自测序列都有一定的相似性,从而在“通过blast截取同源序列”的过程中,这些长序列被截取成了不同的片段。

遇到这种原因,则删除多余的重复序列(通常是碱基数小的那一条)。
第二种原因,可能是某些序列的方向和其它序列不一致。通常这种序列在mega软件中显得特别“突兀”。

可以把这条“可疑”序列和其它的“正常序列”中的一条上传到Genbank中相互Blast后,验证方向是否一致。

Blast后,核对两条序列起止数字是否都是终点大于起点,如果不都是终点大于起点,说明方向反了。
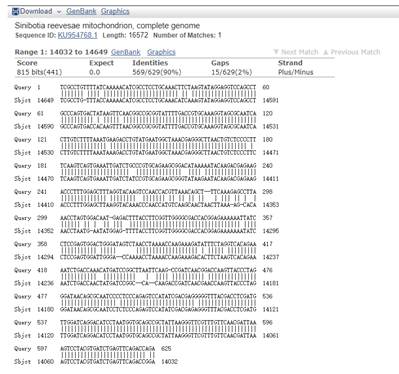
对于这样的情况,需要将可以序列进行“反向互补操作”纠正其方向,才能进行后续分析。
操作方法是,在mega中选中“可疑序列”,在“Data”菜单栏中选择“Reverse complement”。

https://m.sciencenet.cn/blog-508298-1169764.html
上一篇:每日翻译20190228
下一篇:每日翻译20190330
扫一扫,分享此博文
全部作者的精选博文
全部作者的其他最新博文
全部精选博文导读
相关博文
- • Minerals线下恳谈会:履践致远、与时偕行——对话中国科学院广州地球化学研究所期刊合作学者
- • 聚英才 建高地 | 北京理工大学“特立青年学者”全球招聘开启
- • 700年后日本或濒临灭绝?日本学者推算预测:届时或仅剩1名15岁以下孩子
- • [转载]【同位素视角】非英语母语学者如何区分’e.g.’, ‘i.e.’, ‘namely’与‘such as’等混淆难题
- • 美国佐治亚大学等机构学者:刈割策略对Bulldog 805紫花苜蓿+Tifton 85狗牙根混播草地产量及品质的影响
- • 美国堪萨斯州立大学、密苏里大学等机构学者研究成果:土壤水分管理策略和品种多样性对紫花苜蓿产量、营养品质和农场盈利能力的影