博文
我们不知道答案的125个科学问题(66)基因组规模
 精选
精选
||
66. 为什么有些生物的基因组很庞大而另一些却很小?
Why are some genomes really big and others quite compact?
地球上存在着各种各样的生物,而这些生物都是如何保持它们千差万别的特征和形态的?这就是生物遗传过程中的基因组问题,本问题将从基因组的角度分析保持物种特征的基因组为什么有的很庞大而有些却很小?
1. 基因组的概念
任何动植物都可以从一个细胞发育而来的事实让人们相信,在细胞之中一定保存着某种让动植物成长发育成不同个体的完整信息。进一步,植物体细胞可以培养成整株植物体和动物细胞通过细胞核移值(将体细胞的核替换卵细胞的细胞核)可以克隆出动物个体这两件事实让生物学家进一步确信:让一个细胞发育成个体的信息一定存在于细胞核中。经过生物学家们不断的努力最终确定:生物个体成长的全部信息都保存在细胞核内一种被称为染色体的DNA中,这些具有双链结构的DNA分子内部有四种碱基对的有序排列包含着生物体从一个细胞发育成完整个体并完成出生到死亡整个过程的密码信息。现在科学家就将生物细胞核中DNA上存储的所有指令密码总称为生物的基因组(genome)。所以,基因组是细胞中一整套DNA指令的总和,它是生物体中完整遗传信息的集合。

图1 基因组、染色体和基因的示意图
生物体基因组的信息被储存于染色体的DNA分子链中,而DNA中的一部分或某个片段编码了生物体所需的某种RNA分子和特定的蛋白质分子,就被称为一个基因(注意基因是DNA的一个片段,它提供制造特定蛋白质的指令,然后对应执行特定的功能),所以DNA可以看成是由一段一段的基因组成的序列,从这个意义上生物的基因组就是由这一组组基因的序列构成的,或者可以说生物体所有DNA基因的集合就被称为基因组(见图1所示)。对人类而言,几乎所有的人都有排列顺序大致相同的基因组,也就说任何人都有超过99.9%的DNA序列或基因组与其他任何人相同。
在真核生物中,每个细胞的基因组都包含在一个被内膜所包裹的细胞核中,而原核生物没有内膜,它们将基因组储存在细胞质中称为类核的区域。另外对动物(植物)细胞,还有一些基因在线粒体(叶绿体)中,而这些基因也应包含到基因组中。由此,根据基因组的概念,相应的基因组表达的全部RNA分子就被称为转录组,基因组产生的全部蛋白质就被称为蛋白质组,而研究和分析基因组的学科就被称为基因组学(genomics)。
2. 基因组的大小
明确了基因组的概念,下面我们来看基因组的规模或大小(size),也就是基因组所对应的信息量的多少。一个最为直接的衡量DNA信息量的方法就是计算DNA链中碱基对(base pair: bp)的个数,如1 k bp就表示1000个碱基对。当然为了方便也用DNA的质量来表示基因组的大小,如皮克:1 pg=10-12 g。然而我们知道正常细胞的染色体由两条基因序列相同的染色单体构成,所以基因组的大小一般采用单倍体染色体(比如经过减数分裂的配子细胞的染色体组)中DNA的碱基对数或质量数来表示,这个值就被称为基因组的C值(C-value),其单位可采用皮克pg或兆碱基对数Mbp(1 pg ≈ 10亿bp=1000 Mbp)。C值的意思就是常数(constant),指的是在给定的生物体或物种中,其基因组的大小在细胞之间是恒定的。所以通常的C值都是指单倍体染色体的基因组大小,而对于多倍体的个体、群体或物种它们存在两套以上的完整染色体,此时如何表达基因组大小尚无普遍共识,主要原因是这两套以上的染色体仅仅是简单的重复还是在不同的副本之间会隐藏着一些不可忽略的遗传信息。所以在多倍体生物中,采用单倍体染色体DNA基因组的C值可能只占细胞中DNA总量的三分之一或四分之一或更少,采用单倍体染色体C值会丢失多倍体物种某些具有重要表型相关的DNA信息,有些生物遗传学家则建议将基因组大小和C值解耦,或采用多个C值,如1C-value指单倍体基因组大小,2C-value(或2C)指二倍体的C值等等。
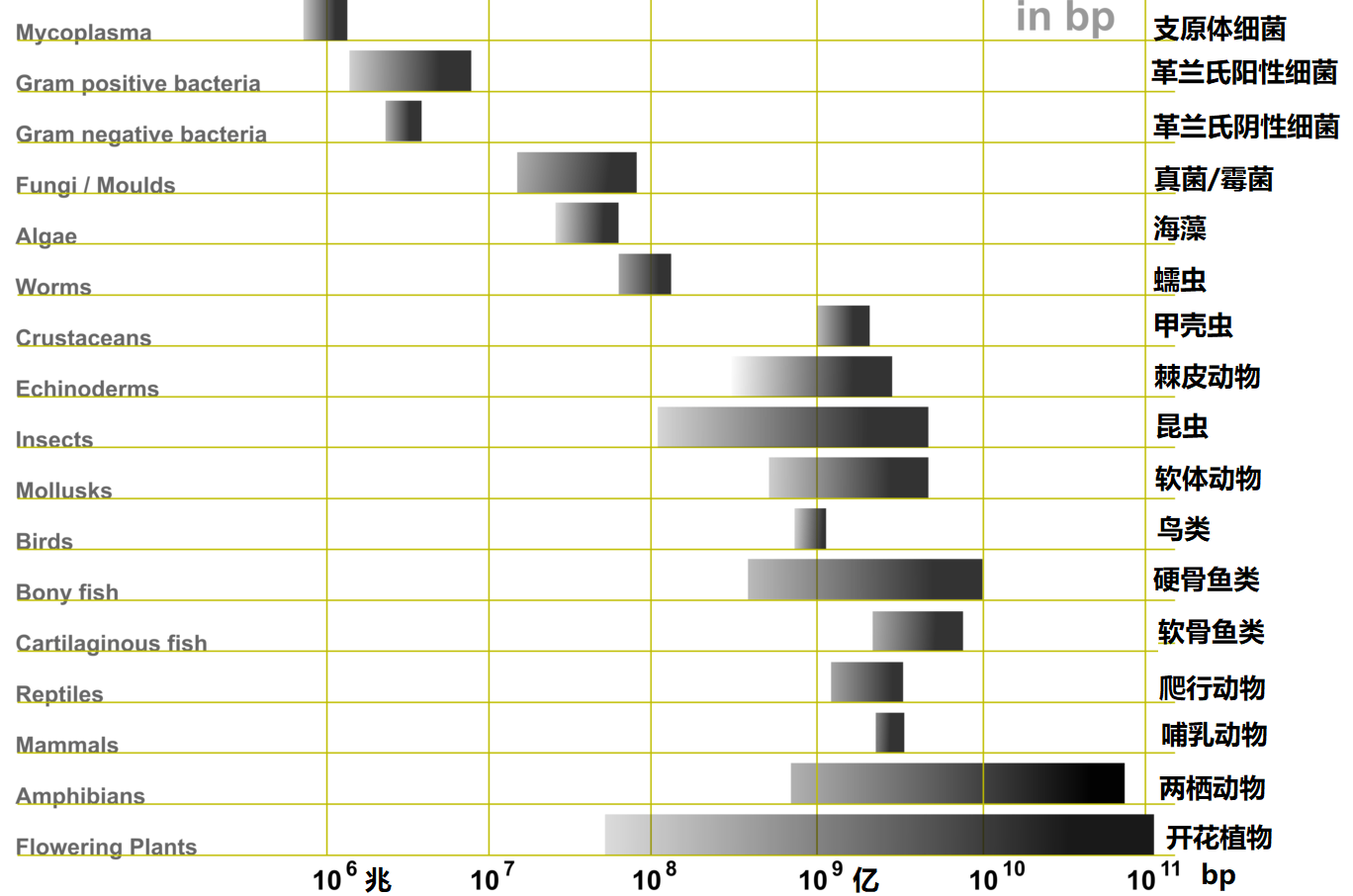
图2 不同生物基因组数据的范围及比较
利用通常意义下的C值(单倍体染色体基因组的规模),生物学家对各种生物的基因组数据进行了广泛的研究和比较,发现了一些令人奇怪的现象(如图2所示)。首先,生物基因组C值的变化范围非常广,有些物种的基因组小得令人难以置信,比如病毒和细菌的基因组,而另一些基因组却大得让人无法理解,比如在一些植物中发现的基因组。但生物整体的C数据基本上可以显示:随着生物复杂性的增加,从原核生物到真核生物以及从无脊椎动物、脊椎动物、植物,物种的C值基本上在普遍增加。然而一些非常相近的生物在C值上却存在有很大的差异(例如两栖动物),而对于诸如人类这种复杂的哺乳动物来说,其基因组C值大约只有3 × 109 bp (30亿bp)。人类的基因组包含的大约30亿个核苷酸序列虽然很大,但日本一种名为“衣笠草”(Paris japonica)的罕见花草的基因组大小约为1500亿bp,竟然是人类基因组的50倍(如图3所示,衣笠草有40条染色体,是由四个异源种的多倍体杂交后的八倍体植物,拥有世界上所有生物体中已知最长的基因组:约1490亿个碱基对),而且地球上还有许多物种的基因组都要比人类基因组大得多。这些奇怪的基因组数据表明生物的复杂性和基因组大小之间似乎没有相关性,这就是著名的基因组C值悖论(C-value paradox,1971年由遗传学家Thomas提出)。换句话说,存在于真核生物中的C值悖论意味着更大的基因组并不会导致更复杂和更高级的物种。

图3 衣笠草照片
3. 基因组差异的原因
基因组C值悖论让我们必须重新认识DNA碱基对序列数和基因的关系。首先,真核生物基因组中基因的数量会远远小于编码蛋白质所需的数量。比如,哺乳动物只有3万到5万个基因(哺乳动物一个基因的平均大小为3000个bp),但它们的基因组大小(或C值)却是30亿bp。虽然人具有99.9%相同的基因组,但显然人种之间是非常不同的,平均而言,一个人类的基因会有1-3个不同的碱基(1-3bp),但这些小差异却足以改变蛋白质的形状和功能,改变蛋白质的合成量、合成时间和合成地点等等,所以它们会影响人的眼睛、头发和皮肤的颜色。更重要的是,基因组的个别变化会极大地影响个体患病的风险和对药物的反应。虽然基因组大小对某种生物保持稳定,但基因组大小在生物演化中会通过基因突变或变异(mutation)而发生改变。图4显示的是基因的3种常见的变异方式,其中第1种删除或插入(delete/insert)和第2种复制(segmental duplications)都会改变基因组的大小,而第3种基因转位(inversion)虽然并不改变基因组的大小,但对生物的表征往往会造成重大影响。

图4 基因的变异和基因组大小的演变
由此可见,生物的复杂性表征和基因组大小的不一致至少可以揭示以下几个基本事实:(1) 基因组中基因编码序列并非是同等重要的,比如基因中的内含子和外显子序列显然在基因蛋白质表达中具有不同的地位;(2) 基因组中某些基因的序列不仅仅只决定于固定的编码序列信息,还决定于基因的调控因子,不同的调控因子在同样的编码序列上会造成非常复杂的不同表达;(3) 基因组中还存在某些不起作用的基因序列,称为伪基因或假基因,基因多备份的冗余信息,基因与基因之间的间区序列(intergenic sequences)以及基因组中高度重复的部分(repetitive DNA) 等等都会增大基因组的C值。以上这些原因都将导致基因组C数和生物复杂性的不一致性,基因组中碱基对序列的不同权重、调控策略和无效冗余或重复等都会让基因组规模和蛋白质表征之间产生不对等的对应关系。而且在科学问题3基因的数目中我们已经知道,在蛋白质合成中还存在一种可选择性剪切的基因表达过程,基因可以通过排列组合来生成更多种类的蛋白质,所以基因组大小的C值数据并不能给出生物差异或复杂性的有效表征,这就是为什么有些生物的基因组这么大而有些却非常小的原因之一。
4. 基因组的DNA测序
基因组信息或碱基核苷酸序列要通过DNA测序(DNA sequencing)来获得(比如新冠病毒的核酸序列检测)。DNA测序就是用于确定DNA链中碱基排列顺序的方法,就是能够将DNA碱基信息读取出来的一种测序方法。目前DNA测序主要有两类:一是化学反应标记方法,即桑格测序法(Sanger sequencing)和高通量下一代测序方法(the next-generation sequencing),另一种是微观分子测序法或者被称为非桑格法的第三代分子测序方法。
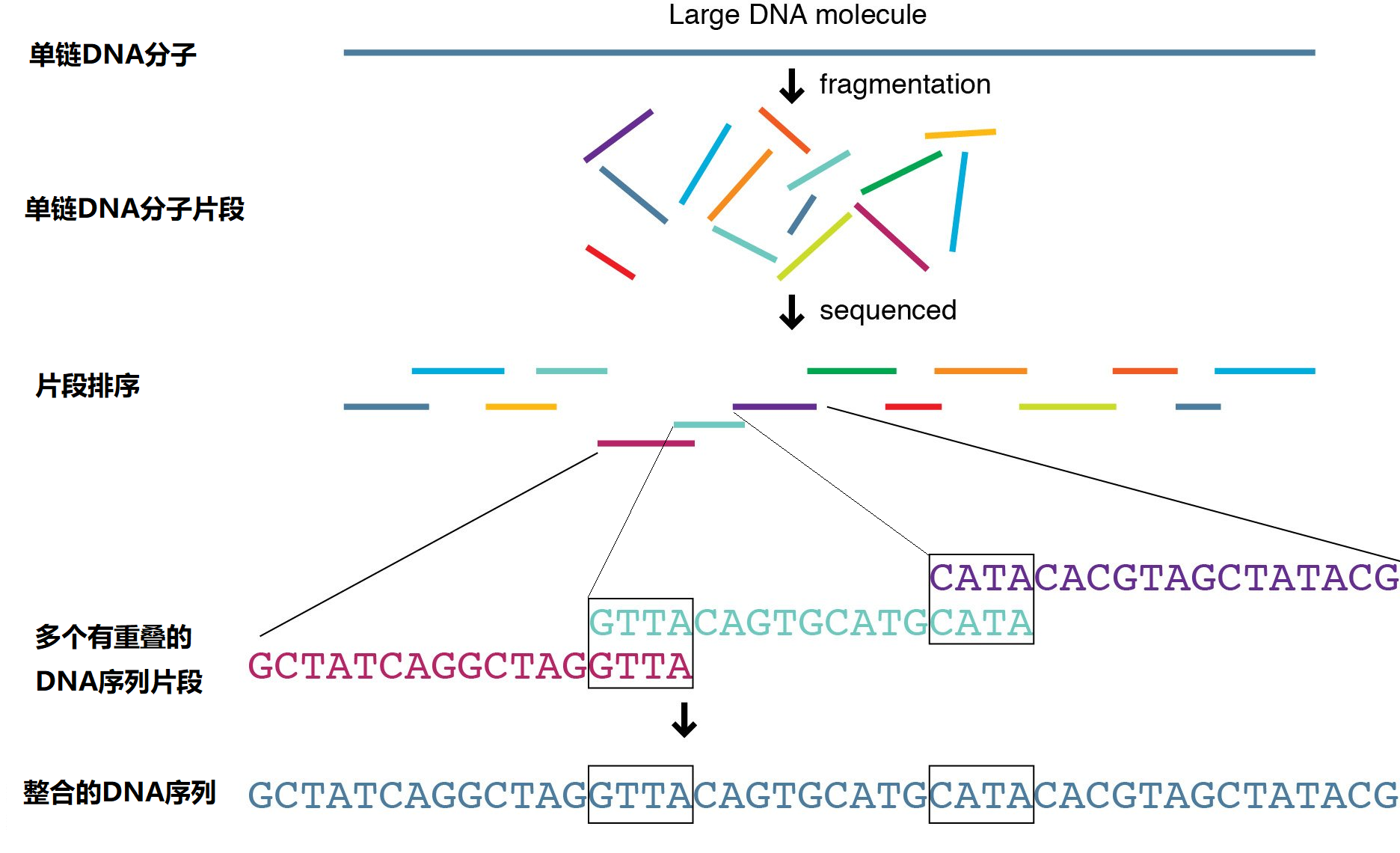
图5 DNA测序处理过程示意图
桑格测序由弗雷德里克·桑格(Frederick Sanger)于1975年发明,因为对DNA序列解析等方面的贡献两次获得诺贝尔化学奖。在桑格测序中(见图5所示),先将待测的DNA链打成一段段的DNA片段(比如600bp长度的片段,可以使用剪切酶准确切断或其他机械方法随机打断),然后再对筛选的一某段DNA链进行预处理(包括提纯、扩增、高温单链化等)。接着如图6所示,先在待测DNA片段中加入前趋物进行起始标记(便于序列定位),然后依次进行四个单独的DNA合成反应,这四种反应都包括正常的N = A、G、C和T的脱氧核苷酸(dNTPs)和DNA聚合酶(用来合成DNA的酶),并且每种反应都只加入四种双脱氧核苷酸三磷酸(ddNTPs)中的一种:ddATP、ddGTP、ddCTP或ddTTP,这四种ddNTPs加入哪一种,DNA反应就会终止于哪一个N碱基上,所以可以分别命名为A、G、C和T反应(当ddNTP与核苷酸链结合时,DNA合成会终止是因为ddNTP分子戊糖3'上缺少一个的羟基,见图6下方插图,这是与链中的下一个核苷酸形成连接所必需的)。由于ddNTPs是随机结合的,因此每个反应的DNA分子合成会终止于各自不同的碱基位置。合成不同长度DNA双链后,经过A、G、C和T反应的产物(经过高温单链化并去除没有反应的核苷酸)被分别装入充满凝胶的四个通道中,使用凝胶电泳(合成DNA分子片段越小运动越块)进行分离。检测凝胶的DNA沉积条带,然后从凝胶的底部到顶部读取序列,包括所有四个通道的条带。例如,如果所有四条通道中最低的条带出现在C反应通道中,则序列中的第一个核苷酸就是C。如果从下往上的下一个条带出现在G反应通道中,则序列中的第二个核苷酸就是G,依此类推。由于在反应中使用了双脱氧核苷酸ddNTPs,所以Sanger测序也被称为“双脱氧”终止法测序。
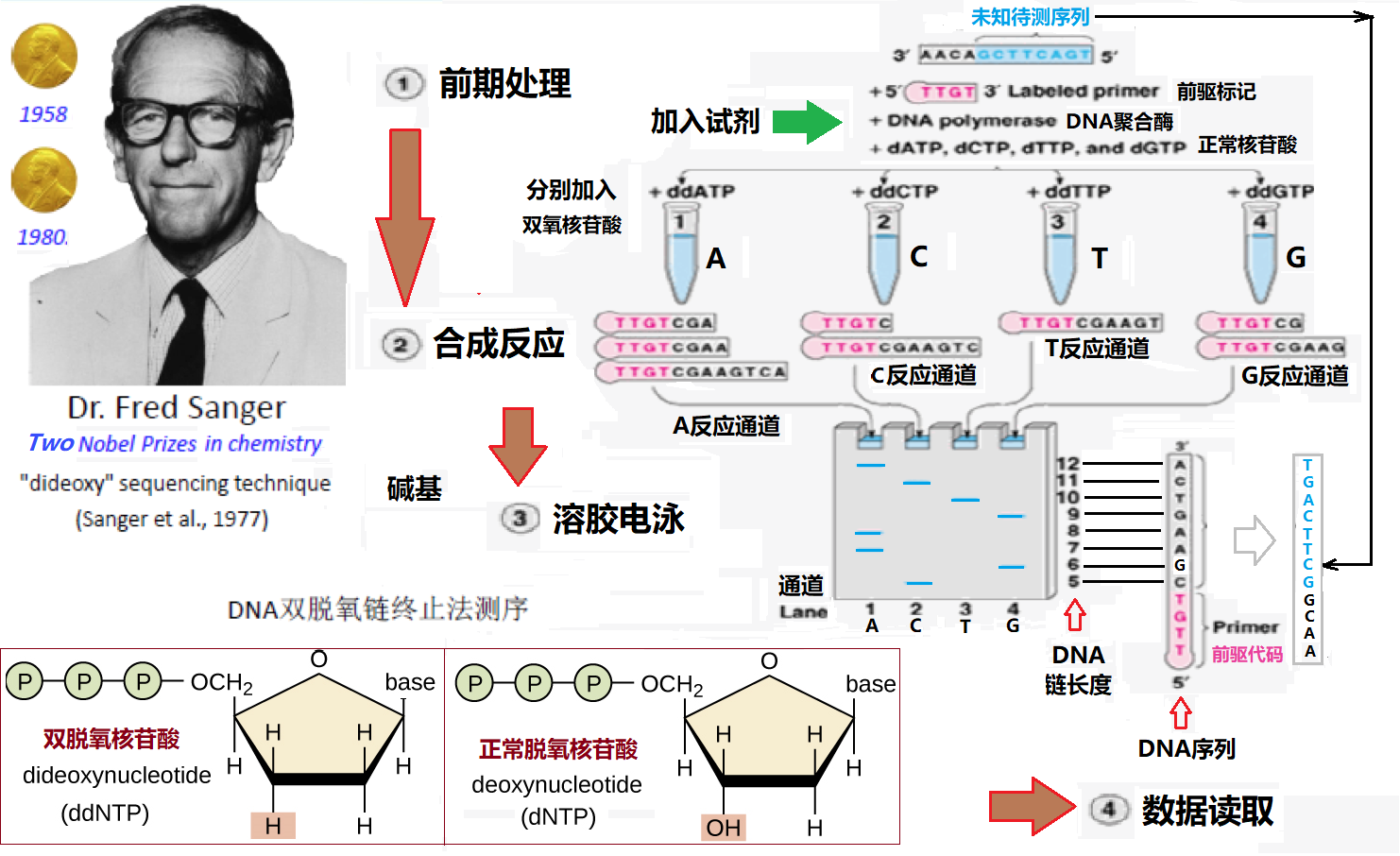
图6 桑格测序方法示意图
当然上面讲的是传统的格桑测序方法,而现在使用的下一代测序方法是在桑格法上发展而来的,首先是将ddNTPs采用不同的荧光标记,将电泳沉积改为荧光检测法(采用毛细管凝胶电泳,越短的DNA片段会先到达检测点被激光激发记录,不同荧光颜色对应不同碱基,见图7)。为提高检测速度,又发展了边合成边测序方法,在合成测序反应中,使用不同的荧光颜色标记不同的核苷酸dNTPs,当DNA聚合酶将带有不同荧光标记的核苷酸链接到DNA链上时,该核苷酸就被光源激发出对应的荧光,荧光信号则被检测器检测并利用计算机自动翻译输出。这种方法可以连续产生125个核苷酸的“读取”过程,一次可以产生数十亿次的读取。为了组装一大块DNA中所有碱基的序列,研究人员需要读取重叠片段的序列。这样较长的序列可以由较短的片段组合而成,有点像把一个线性拼图拼在一起。在这个过程中,每个碱基不仅要读取一次,而且要在重叠的片段中至少读取几次,以确保准确性。

图7 荧光测序的不同碱基序列读取的荧光信号
第三代分子层次上的DNA测序就是可以在分子层次有效读取每一个不同核苷酸信息的检测方法,微观方法不仅能准确读取不同碱基上的不同生物化学标记,还可以直接读取碱基的电学、磁学信号等等。比如根据通过不同的核苷酸配对键上的电流信息来进行碱基信息测量的纳米孔测序方法(如图8所示)。当然如果可以直接读取利用光镊拉开的单链DNA上不同碱基的电子云分布信号的话,就可以发展一种直接读取DNA碱基序列的测序方法。

图8 一种DNA纳米孔分子测序方法示意图
无论如何,DNA测序技术的发展主要体现在DNA测序速度和准确率的提升上,当然测序成本的下降将使DNA测序获得更加广泛的应用。总之,DNA测序技术的发展,为基因组的研究获得了更为准确和完整的数据库,也为人类社会带来了更多有用的工具,DNA测序技术在疾病的认识和治疗中越来越发挥更大的作用,对人类基因组的遗传变异、基因突变及单个碱基对的替换、删除或添加等基因组演化提供了更为精准的研究依据。
https://m.sciencenet.cn/blog-318012-1413372.html
上一篇:我们不知道答案的125个科学问题(65)端粒和着丝粒
下一篇:我们不知道答案的125个科学问题(67)垃圾DNA