博文
《自然—化学生物学》:浙江大学刘婷团队揭示BRCA1/2缺陷细胞通过复制叉稳定性重建导致PARP抑制剂耐药的新机制
||
PARP抑制剂利用合成致死(Synthetic Lethality)效应杀死携带BRCA1/2(Breast cancer susceptibility gene)缺陷的肿瘤细胞,在临床上具有重要意义。然而,一部分肿瘤细胞可以通过恢复同源重组修复,或重建复制叉稳定性等机制,对PARP抑制剂产生耐药。由于学术界对复制叉稳定性维持机制的研究尚浅,因此对BRCA1/2缺陷肿瘤细胞如何通过重建复制叉稳定性获得PARP抑制剂耐药性的分子机制也知之甚少。
北京时间2024年4月22日,浙江大学基础医学院刘婷课题组在Nature Chemical Biology杂志在线发表题为“UFL1 triggers replication fork degradation by MRE11 in BRCA1/2-deficient cells”的最新研究成果。
该研究发现,在复制压力下,E3连接酶UFL1催化PTIP蛋白发生UFMylation修饰,促进BRCA1/2缺陷细胞中新生DNA链的降解。这一分子机制表明,BRCA1/2缺陷细胞可以通过失活UFMylation通路来重建复制叉稳定性,从而对PARP抑制剂产生耐药。这也是国际上首次报道UFMylation参与复制叉稳定性维持的功能与分子机制。
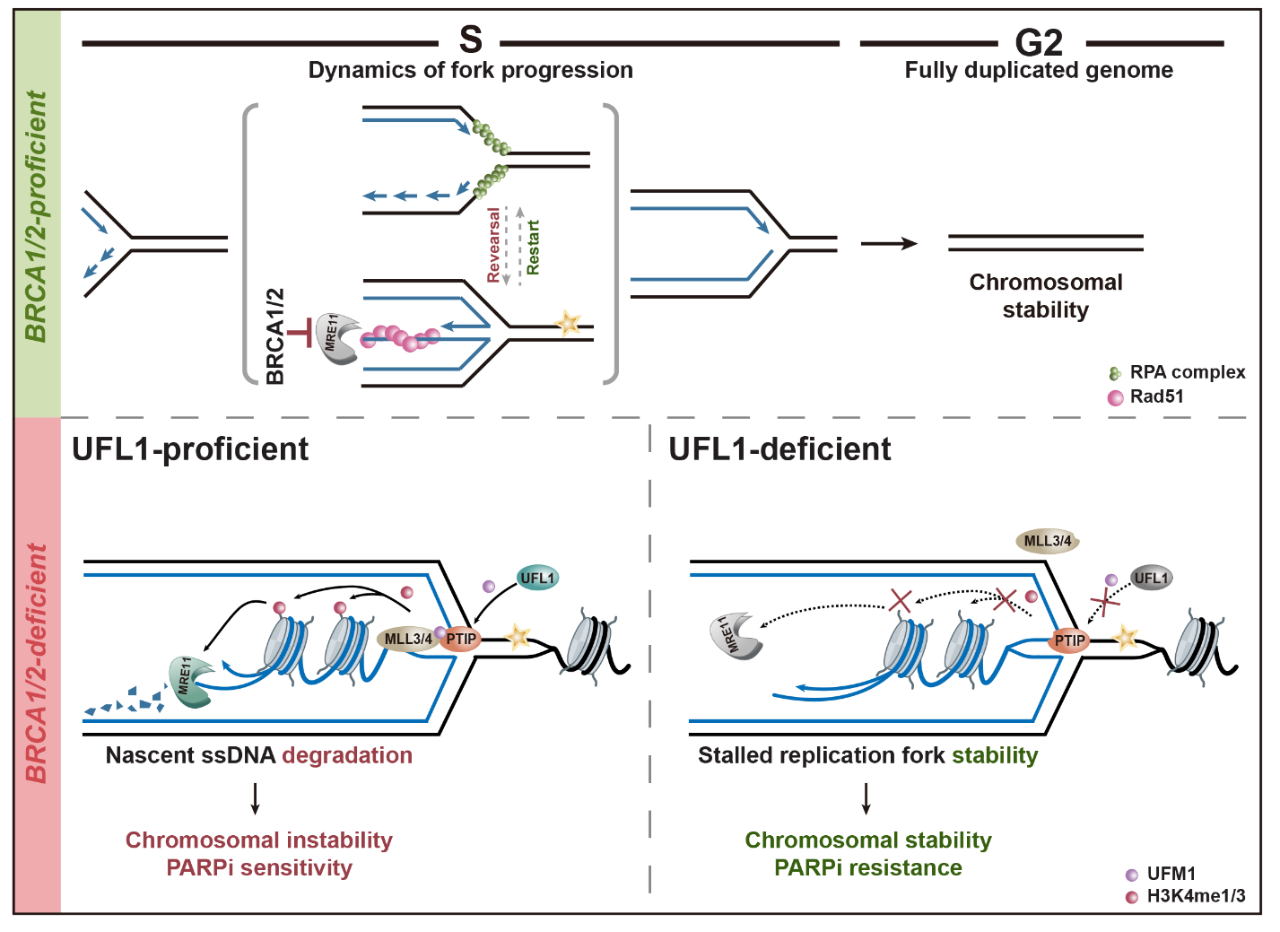
复制压力发生时,解旋酶与聚合酶发生解偶联或受到核酸酶修剪,导致停滞的复制叉暴露出单链DNA(ssDNA),并与单链结合蛋白RPA复合体结合;随后,重组酶RAD51置换RPA,形成ssDNA-RAD51复合物。接着,在转位酶(Translocase)如SMARCAL1、ZRANB3、HLTF和PICH,以及DNA拓扑异构酶TOP2A和SUMO E3连接酶ZATT等的协作催化下,新生子链DNA经过两步翻转并退火。此时,复制叉形成四叉的霍利迪交叉(Holliday Junction)结构,从而暂时稳定了停滞复制叉。该过程中,RAD51促进复制叉翻转的过程,与解旋酶BLM所推动的复制叉重启相拮抗。刘婷课题组于2024年2月在Nature Communications上发表的研究论文报道了TFIP11蛋白结合到停滞复制叉上,通过竞争性抑制BLM与复制叉的结合,促使RAD51与复制叉结合,并促进复制叉翻转的分子生物学机制。此外,为了维持复制的稳定性,停滞复制叉必须防止核酸内切酶SLX4复合物被过度招募其上,切割复制叉并导致复制叉崩塌。刘婷课题组于2024年4月在The EMBO Journal上发表的研究论文报道了ssDNA-RPA复合物所招募的ATR激酶如何阻止SLX4核酸酶复合体的过度募集,从而避免复制叉崩塌的分子机制。
BRCA1/2蛋白在复制叉稳定性维持中也发挥关键作用。当翻转发生时,重组酶RAD51与翻转链的结合需要被BRCA1/2稳定,以保护新生DNA链免受MRE11等核酸酶的降解。在BRCA1/2缺陷的肿瘤细胞中,由于复制叉无法维持其稳定性,这些细胞对PARP抑制剂表现出高度敏感,导致合成致死。
UFMylation是一种类泛素化修饰,其修饰遵循与泛素化修饰相似的E1-E2-E3级联反应过程,将类泛素蛋白UFM1(Ubiquitin-fold modifier 1)共价连接到底物蛋白的赖氨酸残基(K)上,其中UFL1(UFMylation E3-like ligase 1)是目前唯一已知的E3连接酶。自2004年发现UFMylation以来,尽管这一翻译后修饰已被证明在调节胚胎发育、内质网稳态和基因组完整性等方面具有重要意义,但迄今为止,已发现的UFMylation底物十分有限,此前尚未有过UFMylation参与复制叉稳定性调控的报道。
刘婷课题组在本研究中发现,在BRCA1/2缺陷的细胞中,当UFMylation的E3连接酶UFL1被敲减时,部分恢复了由PARP抑制剂所引起的基因组不稳定性,并证明该表型的出现并非由于同源重组修复被重建,而是由于在复制压力下,新生DNA链不再受到降解。进一步的研究表明,在BRCA1/2缺陷的细胞内,UFL1催化核酸酶MRE11的关键招募蛋白PTIP发生第148位赖氨酸(K148)的UFMylation修饰。而PTIP蛋白的UFMylation修饰则进一步促进了PTIP- MLL3/4甲基转移酶复合体的组装,进而使得停滞复制叉处组蛋白H3K4的甲基化水平显著增加。核酸酶MRE11随后响应该甲基化信号,被招募到停滞复制叉上,降解新生DNA。
综上所述,本文首次报道了UFMylation参与调节复制叉稳定性的功能与机制;系统阐明了UFMylation促进BRCA1/2缺陷细胞中新生DNA链在复制压力下发生降解的分子生物学过程;并深入揭示了当UFL1缺失时,核酸酶MRE11无法降解新生DNA链,从而重建复制叉稳定性,使细胞对PARP抑制剂产生耐药性的分子机制。据悉,深圳大学许兴智教授团队在PNAS上也将报道UFL1通过催化PARP1的UFMylation,从而调控复制叉稳定性维持。
该工作主要由研究助理田甜完成。田甜为该论文第一作者,浙江大学基础医学院刘婷教授为该论文通讯作者。本研究得到了国家自然科学基金委、科技部重点研发计划等项目资助。
相关论文信息:
https://www.nature.com/articles/s41589-024-01611-7
刘婷课题组致力于研究DNA损伤应答与基因组不稳定性的分子机制研究,①应用细胞分子生物学、生物化学、生物信息学、结构生物学等多种实验手段,阐明细胞应答DNA损伤、维持基因组稳定性的分子机制;②应用基础研究与疾病模型相结合的方式深入研究与DNA损伤应答异常相关疾病(如肿瘤等)发生的分子机制;③从DNA损伤应答系统入手探寻对肿瘤等相关疾病进行治疗的最佳靶向方式。
研究成果发表于Science、Nat Chem Biol、Nat Commun、Proc Natl Acad Sci U S A、EMBO J、Cell Rep、EMBO Rep.等知名学术刊物,欢迎具有细胞分子生物学等相关背景的学生、博士后、技术员等加盟。
课题组详细信息请见网站https://person.zju.edu.cn/tliu
https://m.sciencenet.cn/blog-3423233-1431115.html
上一篇:PNAS:许兴智/汪兆琦团队合作发表类泛素UFM1修饰维持复制叉稳定的研究成果
下一篇:《细胞》:吴军/杨辉/郭帆/周海波团队在小鼠体内生成大鼠前脑组织