博文
Discovery Studio 分子对接前准备
|
分子对接(molecular docking)是依据配体与受体作用的“锁-钥原理” (lock&key principle),模拟配体小分子与受体生物大分子相互作用的一种技术方法。
在Discovery Studio(DS)进行分子对接我们需要进行如下操作。a. 准备受体;b. 准备配体;c. 定义结合位点。
1. 准备受体。
DS接入PDB数据库,只要输入PDB的ID即可自动下载。File-Open URL 选择PDB Structures,(DS2022接入了AlphaFold Protein Structure Database)。
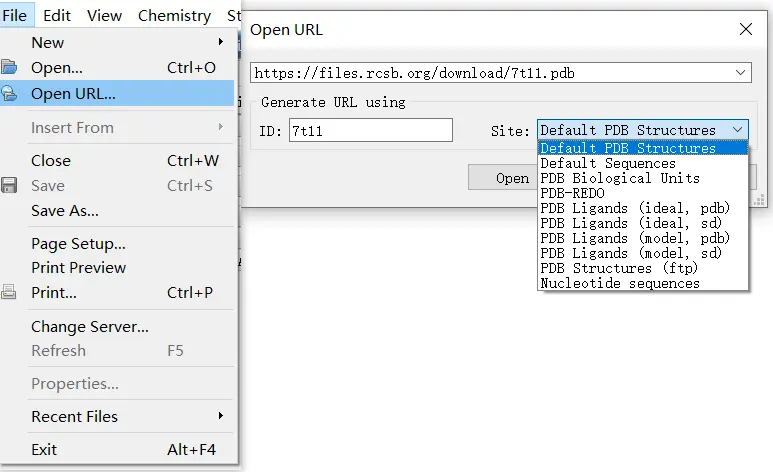
此功能需要联网
也可以选择open 来打开蛋白文件,或者直接当蛋白文件拖入DS中。
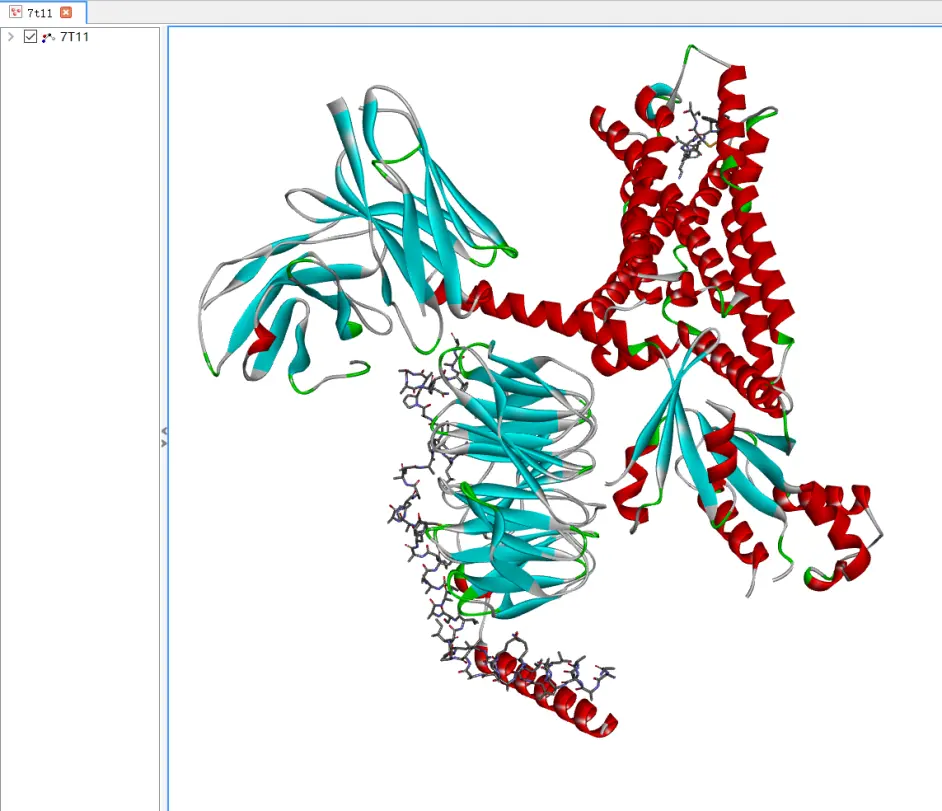
打开的蛋白
然后将该蛋白进行前处理准备,使用大分子模块中Prepare Protein功能,该功能为进一步建模操作准备了导入的蛋白质结构。解决通过实验或第三方建模方法获得的蛋白质结构中常见的问题。
步骤如下(可保持默认):
标准化原子名,在残基中插入缺失的原子,并去除交替构象。也可以根据Advanced|Keep Waters和Advanced|Keep Ligands的设置去除水和配体分子。
根据SEQRES数据或用户指定的循环定义插入缺失的循环区域(可选)。
使用LOOPER算法(可选)对短、中型环路区域进行优化。
最小化剩余的循环区域(可选)。
计算pK和质子化结构(可选)
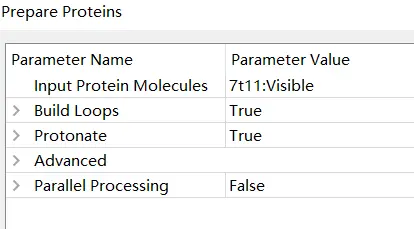
准备蛋白的参数

输出结果:已修复Loops和side-chains
2. 准备小分子
打开方式同上。
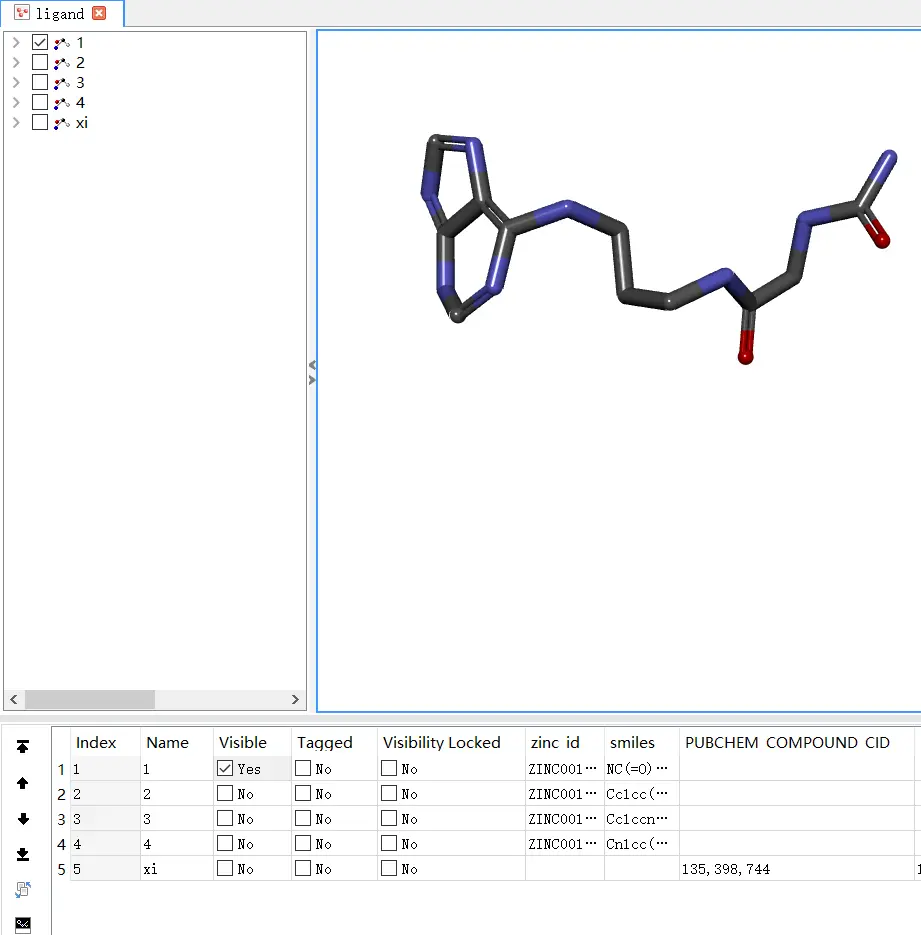
配体未优化
然后打开小分子模块的Prepare Ligands来优化配体,本功能为输入其他协议准备配体。它可以完成移除重复构象、枚举异构体和互变异构体以及生成3D构象等任务。
许多协议(如De Novo Library Generation)需要合理的起始配体结构来获得良好的结果。协议也可能受益于枚举有效的电离态、互变异构体和同分异构体(例如,对接协议)。您还可以通过删除具有不良特性的重复或化合物来节省时间。本协议通过执行以下步骤来实现这一点,其中一些步骤可以通过协议参数来控制:
生成规范互变异构体。
保留最大的碎片。
对常用官能团设定标准形式电荷。
Kekulize分子。
枚举电离状态在给定的pH值范围或设置他们根据预定义的模板(可选)。
列举互变异构体(可选)。
列举同分异构体(可选)。默认情况下,只枚举未指定的键和原子。
删除重复结构(可选)。
违反Lipinski规则的过滤器结构(可选)。
使用Catalyst生成合理的3D构象(可选)。
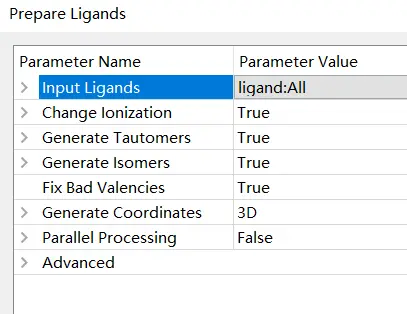
准备配体参数列表
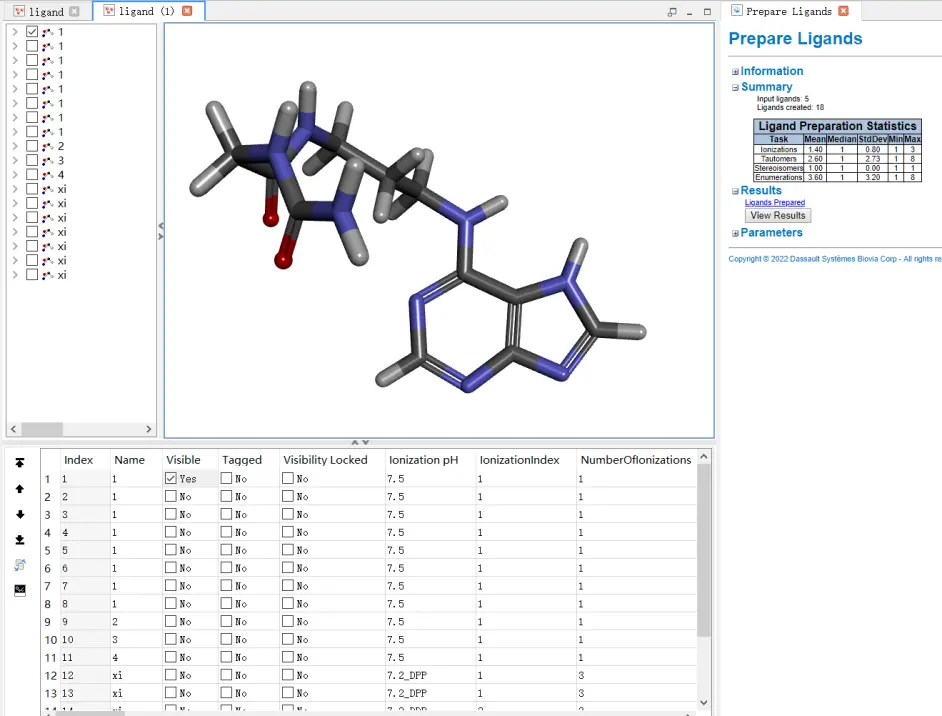
准备配体结果,可列出手性结构
3. 定义结合位点
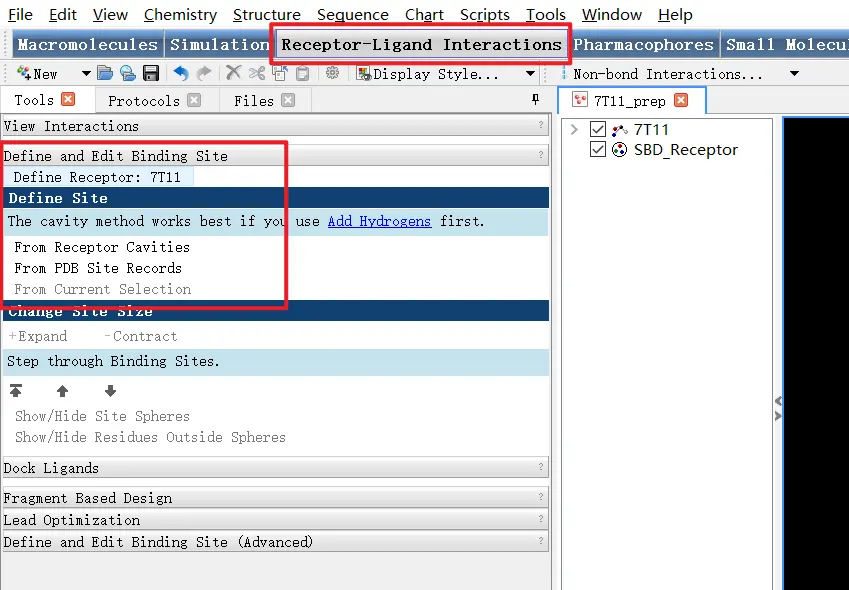
软件位置
DS内置了3种结合位点定义方式:软件预测、PDB记录(从PDB数据库下载的受体才可用)、自定义。
还提供了可以根据已经结合的配体来定义结合位点。
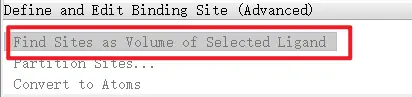
这个默认不显示,可以右上角搜索Find sites找到。
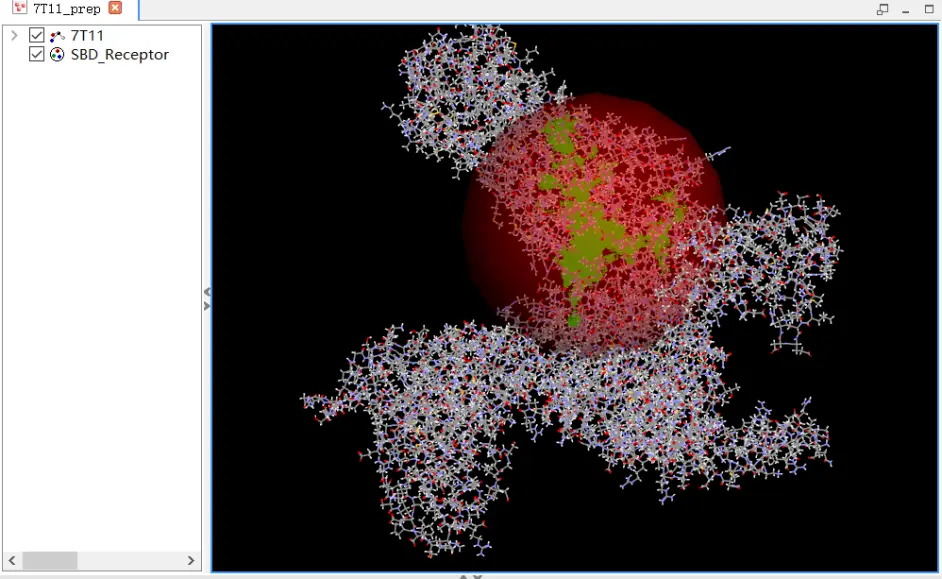
定义受体的活性腔
至此,分子对接前处理准备动作都以妥善,接下来只需执行分子对接程序即可。 DS提供了一系列的分子对接功能,待后期娓娓道来。如下:
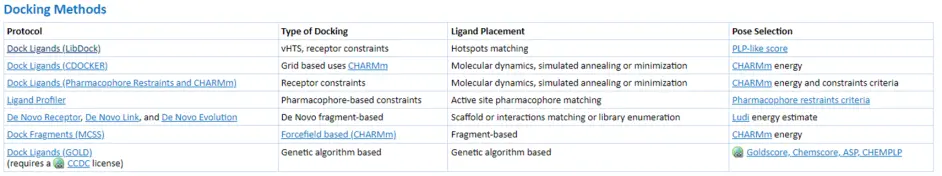
DS内置的分子对接程序

https://m.sciencenet.cn/blog-3536821-1361780.html
上一篇:Discovery Studio虚拟筛选
下一篇:Discovery Studio分子对接后分析结果