博文
Discovery Studio官方教程(Help-Tutorials) 可视化分子对接非键作用
|
所需功能和模块:Discovery Studio Client
所需数据文件:3pwh_dockedposes.dsv
所需时间:20分钟
介绍
分子之间的非键相互作用是生命体系中分子识别的基础。在基于结构的药物设计过程中,识别和优化配体分子和受体分子间的相互作用是一个基本过程,因此,全面细致的分析非键相互作用非常关键。
教程将采用热稳定的腺苷A2A受体和反相激动剂ZM241385的复合物,其中A2A受体是G-蛋白偶联受体,是帕金森氏症的药物靶标,其拮抗剂Preladenant正处于临床阶段。本教程将分析对接至A2A受体的配体分子构象。
执行计算并分析结果
在文件浏览器(Files Explorer)中,双击打开Samples|Tutorials|Receptor-Ligand Interactions|3pwh_dockedposes.dsv。
打开一个分子对接结果,包含3pwh蛋白分子和多个小分子的对接构象。(图1)
说明:对接分子共有50个,具有一定多样性,其中20个为参照分子,带有CHEMBL前缀,30个为诱饵分子,带有ZINC前缀。这50个分子基于Dock Ligands (CDOCKER)模块对接至已经预处理过的腺苷A2A受体分子,最终得到3pwh_dockedposes.dsv文件中的对接结果。
本教程通过对该对接结果的分析来获取重要的非键相互作用信息,从而进一步优化虚筛流程。
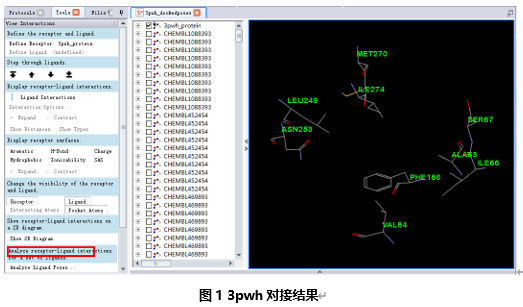
在工具浏览器(Tools Explorers)中,展开Receptor-Ligand Interactions |View Interactions,点击Analyze Ligand Poses。
相应的参数出现在参数浏览器中。
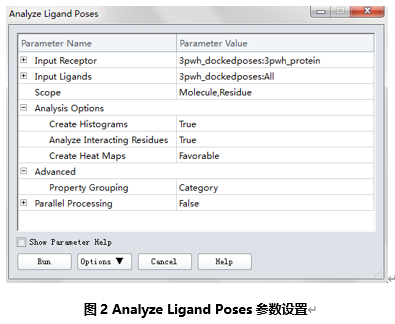
点击Input Ligands参数项,下拉列表中选择3pwh_dockedposes:All,选中3pwh_dockedposes窗口中所有配体构象。
其余参数保留默认设置,点击Run运行任务。(图2)
点击Background让任务后台运行,等待任务完成。
在任务浏览器(Jobs Explorer)中,双击刚运行完的任务,打开Report界面。
在Report界面的Results一栏下,点击View Interaction Histogram(Residues)链接。
打开一个新的界面,该界面中包含了几幅柱状图。
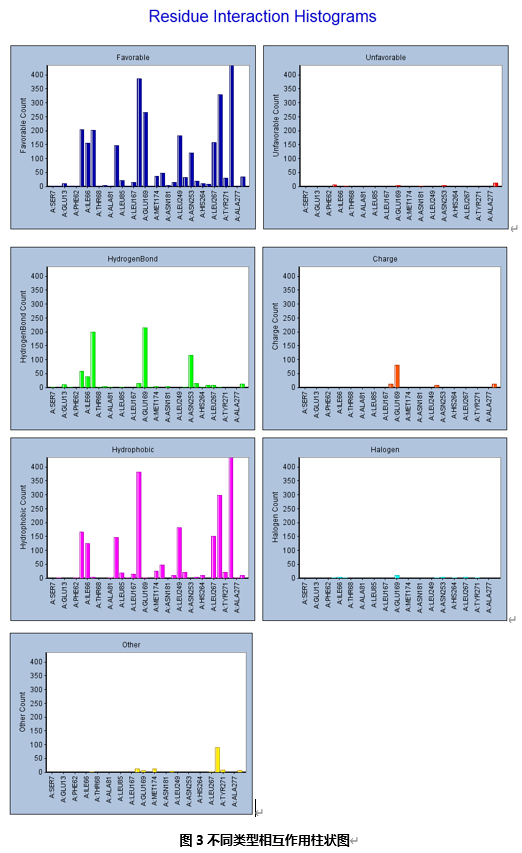
以上柱状图总结了各对接构象同受体之间不同类型的相互作用力。对于每一种相互作用类型,从相应的柱状图可以看出受体蛋白中哪一个氨基酸残基同所有的对接构象形成的该类型作用数目最多。所有柱状图的横坐标都相同,便于进行不同类型作用的比较。
将鼠标放置在任一柱状之上,就会显示同该残基形成的该类型相互作用的配体对接构象数目。
点击 (通过在DS菜单栏下方点击鼠标右键勾选Navigation调出)中的Go Back
(通过在DS菜单栏下方点击鼠标右键勾选Navigation调出)中的Go Back  按钮回到Report界面。
按钮回到Report界面。
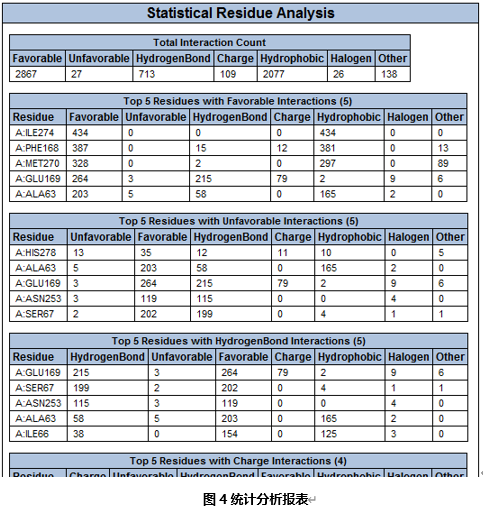
点击View Statistical Information链接。
打开一个新的界面,该界面中包含了氨基酸残基的统计分析报告,该报告总结了相互作用数目及每一种作用类型所涉及的作用数目较多的氨基酸残基。
点击 中的Go Back
中的Go Back  按钮回到Report界面。
按钮回到Report界面。
点击View Heat Map(Favorable)链接。
打开一个个分子窗口和图形窗口,分子窗口中包含了所有的共500个对接构象及相应的非键相互作用信息,图形窗口中含有一张相互作用热图(图5)。
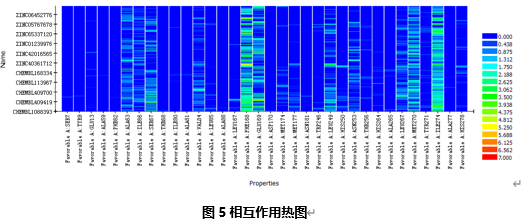
从该图中可以观察到所有参与了同配体之间非键相互作用的残基信息,如PHE168、ILE274。
在打开的分子窗口中,在表格视图中点击鼠标右键,选择Filter。
在弹出的窗口中设置-CDOCKER_INTERACTION_ENERGY的Choice为Top(#),Value为1。
设置Apply filter to each group of identical values选项为Name,点击OK。
该分子窗口中分子会自动按上述设定条件进行过滤,最终每个对接分子只保留-CDOCKER_INTERACTION_ENERGY最高的一个对接构象,共50个对接构象。
注:-CDOCKER_INTERACTION_ENERGY属性已通过事先的ROC曲线分析被证实能够较好的区分活性和非活性分子。
展开菜单栏Chart,点击Heap Map。
在弹出的窗口中设置Y轴为Name,X轴为Hydrophobic A:TYR9,并SHIFT选中HydrogenBond A:HIS278,点击OK。
得到一张疏水、氢键相互作用热图(图6)。
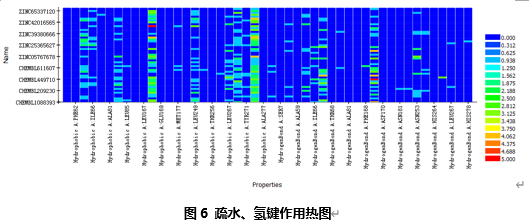
从该热图可以看出所有的参照分子都和ILE274形成了疏水作用,而大部分的诱饵分子也和该残基有疏水作用。而且很难轻易识别出另一种相互作用能够进一步区分参照分子和诱饵分子。然而通过接下来的操作,可以发现采用ILE274和另一种非键作用可以将虚筛结果提高。
切换回filter之后的分子窗口,选中所有50个对接构象,按CTRL+C进行拷贝。
打开一个新的分子窗口,按CTRL+V进行粘贴。
点击鼠标右键,选择Filter。
在弹出的窗口中设置Hydrophobic A:ILE274的Choice为Greater than or equal,Value为1;设置HydrogenBond A:SER67的Choice为 Greater than or equal,Value为1。
点击OK。
最终过滤得到19个对接构象,这19个对接构象满足以下条件:是每个配体分子对接结果中-CDOCKER_INTERACTION_ENERGY最高的对接构象,和ILE274可以形成疏水作用,和SER67可以形成氢键作用。
双击表头Control。
其中有13个control分子,6个decoy分子。虚筛结果有所提高,上述标准可以用来处理后续大规模未知活性分子的虚拟筛选。

https://m.sciencenet.cn/blog-3536821-1362110.html
上一篇:Discovery Studio官方教程(Help-Tutorials) 拉伸动力学计算结合自由能
下一篇:Discovery Studio官方教程(Help-Tutorials) 定义蛋白活性位点以进行分子对接