博文
Discovery Studio官方教程(Help-Tutorials) 分子对接前的准备受体和配体
|
目的: 通过此教程,了解并掌握Discovery Studio中一些基本操作。
所需功能和模块:Discovery Studio Visualizer client
所需数据文件:mdm_random.sd和1aq1.pdb。
所需时间:30分钟
介绍
在使用软件进行课题研究前,我们首先应该了解并掌握该软件使用的一些基本操作。为后续的体系处理做好准备工作。这个教程包括:
l 小分子配体准备
l 蛋白文件的处理
小分子配体准备
在Discovery Studio(DS)中,可以直接构建分子结构,也可以将在其它画图软件中画好的结构直接拷贝到DS中,本教程演示如何在DS中构建小分子结构。
1. 调用Sketching功能
从View菜单下,打开Toolbars,选择Sketching。
Toolbars中将显示各种Sketching的工具,这些工具可以用来构建化合物的初始结构。
2. 利用Sketching构建化合物的3D空间构象
打开一个分子显示窗口(Molecule Window),菜单栏File|New|Molecule Window。
注:DS中有四种窗口模式,包括Molecule window(显示分子结构),Protein Sequence Window(显示蛋白序列),Nucleotide Sequence Window(显示核酸序列),Script Window(显示脚本语言),因此我们需要根据载入的文件类型选择窗口。
DS中构建化合物的3D空间构象非常容易,也非常灵活。本教程以以下化合物为示例,以图示的方法演示如何构建化合物的结构。
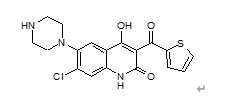
选择 ,在窗口中画出结构1。
,在窗口中画出结构1。
点击 (可以通过菜单栏View|Toolbars|Sketching调出)将其选中,然后选择菜单栏Chemistry|Bond|Aromatic得到结构2。
(可以通过菜单栏View|Toolbars|Sketching调出)将其选中,然后选择菜单栏Chemistry|Bond|Aromatic得到结构2。
选择 ,鼠标指于芳环单键处并单击,构建稠环结构3。
,鼠标指于芳环单键处并单击,构建稠环结构3。
选择 ,构建连接单键,再选择
,构建连接单键,再选择 ,鼠标指于C原子处并单击构建环状结构,最后得到结构4。
,鼠标指于C原子处并单击构建环状结构,最后得到结构4。
选择 和
和 构建单键和环状结构,选择
构建单键和环状结构,选择 再次点击相应的键就可以构建双键结构,最终可得到结构5。
再次点击相应的键就可以构建双键结构,最终可得到结构5。
更换元素类型, 选中某个碳原子,选择菜单栏Chemistry|Element更换相应元素即可,最后得结构6。
选中某个碳原子,选择菜单栏Chemistry|Element更换相应元素即可,最后得结构6。
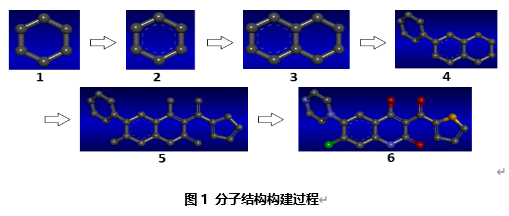
选择菜单栏Structrue|Clean Geometry可进一步优化此化合物的几何三维结构。
3. 使用protocol处理多个配体结构
如果配体数目较多,还要考虑对映异构体等因素时,可用流程浏览器(Protocol Explorer)中的Prepare Ligands处理该体系。
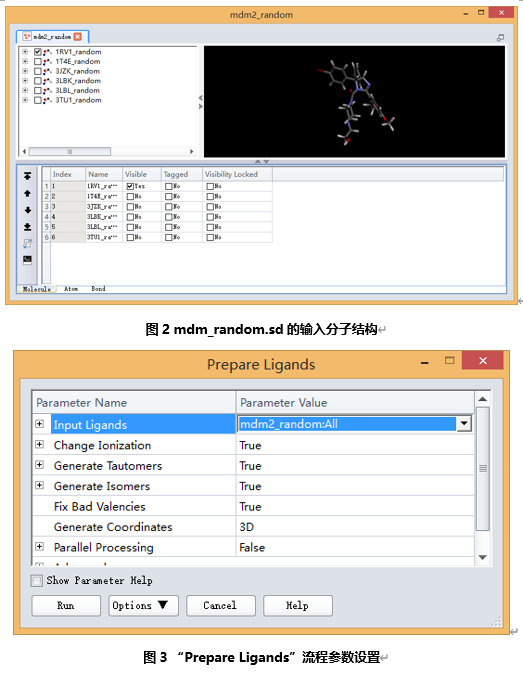
从文件浏览器(Files Explorer)中选择并双击打开Samples|Tutorials|Pharmacophore|mdm_random.sd文件,默认为以表格浏览器(Data Table View)形式打开。将相应分子的表格属性中visible前勾选上,即可观看分子结构(图2)。
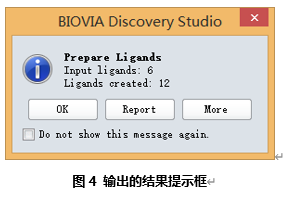
展开工具栏中Small Molecules|Prepare or Filter Ligands,点击Prepare Ligands, 打开相应的流程参数面板(图3)。
点击Run运行作业,等待作业完成。
任务运行完毕,跳出如下提示框。
输入的6个结构,经过处理后共产生12个配体分子。
点击Report,打开report页面。
点击View results观察处理后的分子结构。
从菜单中选择Window | Close All,关闭所有窗口。
注:DS中也可基于一定的力场对小分子结构进行优化,展开工具栏中Small Molecules|Minimize Ligands,点击Full Minimization即可。
蛋白文件的处理
1. 蛋白结构的载入
介绍三种蛋白结构的载入方法。
1.1可通过DS直接从PDB库中下载蛋白结构
点击菜单栏File|Open URL可出现图5对话框,直接输入蛋白ID号。
如果机器联网,即可直接下载到视窗中。
1.2可通过流程浏览器中的RCSB Structrue Search模糊查询所有符合条件的蛋白
展开工具栏Macromolecules|Query Online Databases|Search Databases,点击RCSB Structrue Search按钮, 流程对应参数打开(图6)。
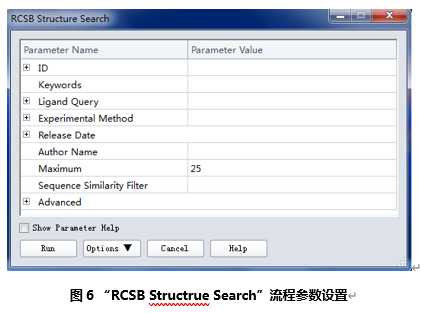
可通过ID号,配体结构信息,实验方法,作者名称等信息查询,可按需要进行检索。
1.3 从File中直接打开本地文件
2. 蛋白文件处理
本例采用第三种载入蛋白的方式,从File|Samples|Tutorials|Receptor-Ligand Interactions文件夹中找到并双击打开1aq1.pdb。
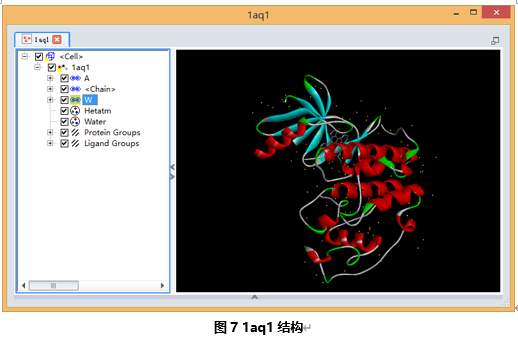
Ctrl+H打开系统视图,Ctrl+T打开表格浏览器,从左边的树状窗口中选择水分子(图7),点击键盘Delete删除。
Clean Protein可去除蛋白多构象,补充非完整的氨基酸残基,为蛋白加氢等,具体参数设置可选择菜单栏Edit>Preference>Protein Utilities>Clean Protein进行设置(图8)。
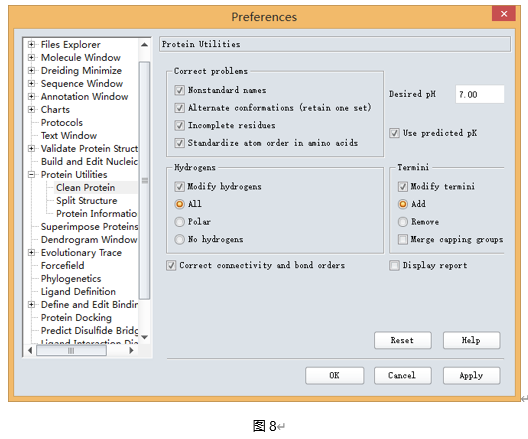
展开工具栏中Macromolecules|Prepare Protein| Mannual Preparation工具面板,点击Clean Protein。
经过处理的蛋白,可进行后续的对接等操作,也可对其显示方式作改变。
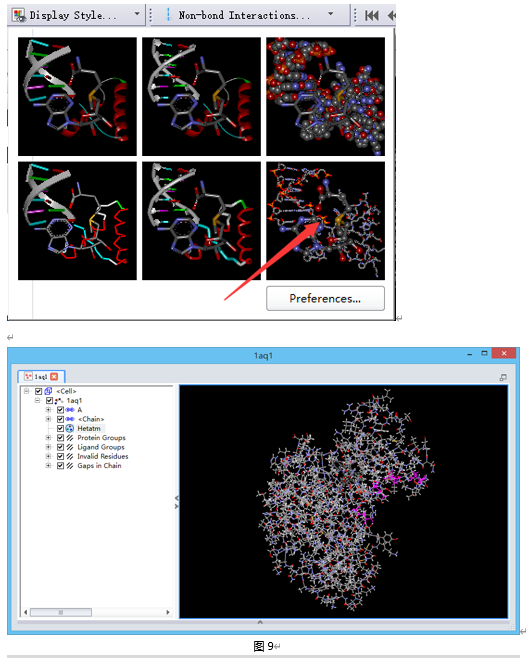
鼠标左键选择Display Style,选择右下角的蛋白质显示方式,蛋白即可按照每个氨基酸残基以线状显示。(图9)
注:此外,也可以通过展开工具栏中Macromolecules|Prepare Protein|Automatic Preparation,点击Prepare Protein对蛋白进行预处理,包括在不同pH条件下的质子化状态,缺失loop的补充等。

https://m.sciencenet.cn/blog-3536821-1362654.html
上一篇:Discovery Studio官方教程(Help-Tutorials) 构建基于受体-配体复合物药效团
下一篇:Discovery Studio官方教程(Help-Tutorials) 创建3D QASR模型