博文
北京雾霾中存在致病菌
 精选
精选
|||
对经受北京咳的人来说,城市严重雾霾的健康危害性再清楚不过。但是具体会产生那些影响并不清楚,或者这些棕色的雾霾是否会加快那些细菌的传播也不明确。

2014年1月(《自然》网站出现错误为2013年),中国学者现在用基因组序列测定技术,确定了北京雾霾中大约1300种不同类型的细菌。不过这些细菌大部分是良性的,只有少数可能会引起过敏和呼吸系统疾病。随着污染程度的加严,这些细菌的数量会增加,提示对人群健康危险有潜在增加,尤其对那些老年人或免疫功能低下的人群。
过去确定微生物一般依赖样品培养,但这种传统方法容易忽略一些关键的细菌类型。过去几年,科学家利用细菌基因组分析确定空气中的细菌类型,这种方法被称为宏基因组学。课题组成员之一,清华大学生物学家Ting Zhu说,这是首次用该方法分析空气颗粒中的细菌类型,这对于评估这些细菌的致病性十分重要。这一研究证明这种方法能够确定细菌种类,对我们理解我们每天呼吸了那些东西十分重要。
研究人员连续7天采集14个空气样本。并对这些空气中的颗粒分成2类,一类是2.5 微米以下PM2.5,另一类是10微米以下PM10。实验期间,北京的PM2.5水平达到500 mg每立方,这个数据超过WHO规定标准的20倍。
先从这些样本中抽提DNA并测序,和大型基因数据库进行比对。结果发现,这些颗粒中细菌的类型主要是隐暗地嗜皮菌Geodermatophilusobscurus,这是土壤中普遍的细菌类型,但也发现存在可导致肺炎的肺炎链球菌,可导致过敏的真菌烟曲霉,大量存在于粪便中的细菌。在雾霾严重的时间,这些DNA的数量可以增加2-4倍。因为数据并不能排除来自已经死亡的细菌,所以不能确定这些细菌是否仍有致病性。应该结合临床患者呼吸道细菌类型的比较,以分析是否由于雾霾的原因。
虽然意大利和美国科学家同行对这个研究比较认可,甚至成为今天《自然》的头条新闻。但我个人认为,这个思路似乎不是那么理想。通过DNA测序,使用先进的细菌基因学技术,确定细菌类型自然似乎一个手段进步(因为这种技术已经在肠道菌群等方面广泛使用,也算不上先进),但确定这些细菌的类型基本都是一种必然结果。设想在没有雾霾的时间,只要采集的样本数量足够多,细菌的类型恐怕也不少,上述所谓致病菌也会存在。问题的关键是,这些随着空气颗粒增加的细菌是否足以导致疾病的发生,到底多少数量才会是雾霾引起健康问题的生物学因素。
虽然不能说这个研究毫无价值,但只分析分析空气中的细菌类型,结果发现随着颗粒增加细菌数量也增加,增加的又都是粪便和土壤中的细菌(水中的细菌类型也是如此),这没有太大医学或卫生学价值,是个无病呻吟的研究项目。如果这个课题来自一个普通的学校和研究机构还凑合,但不幸是来自清华大学这样的一流重点大学。对比日本青年学者建立了非常简单的诱导干细胞的技术,我们这个研究简直就是高射炮打蚊子。关于雾霾的健康危害性显然值得探讨,更重要的是如何控制雾霾的发生,就比如你要研究多少度水会导致烫伤,并不是要让人接受这种危害,这样的研究论文到底何用?希望有人给我一个更满意的解释。
Nature doi:10.1038/nature.2014.14640
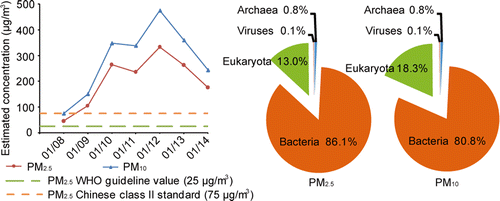
Particulate matter (PM) air pollution poses a formidable public health threat to the city of Beijing. Among the various hazards of PM pollutants, microorganisms in PM2.5 and PM10 are thought to be responsible for various allergies and for the spread of respiratory diseases. While the physical and chemical properties of PM pollutants have been extensively studied, much less is known about the inhalable microorganisms. Most existing data on airborne microbial communities using 16S or 18S rRNA gene sequencing to categorize bacteria or fungi into the family or genus levels do not provide information on their allergenic and pathogenic potentials. Here we employed metagenomic methods to analyze the microbial composition of Beijing’s PM pollutants during a severe January smog event. We show that with sufficient sequencing depth, airborne microbes including bacteria, archaea, fungi, and dsDNA viruses can be identified at the species level. Our results suggested that the majority of the inhalable microorganisms were soil-associated and nonpathogenic to human. Nevertheless, the sequences of several respiratory microbial allergens and pathogens were identified and their relative abundance appeared to have increased with increased concentrations of PM pollution. Our findings may serve as an important reference for environmental scientists, health workers, and city planners.
https://m.sciencenet.cn/blog-41174-763914.html
上一篇:慎重开发氢气相关产品
下一篇:关注内皮细胞的氧化损伤