博文
R语言构建单倍型网络
||||
#编者信息
熊荣川
明湖实验室
xiongrongchuan@126.com
http://blog.sciencenet.cn/u/Bearjazz
library(haplotypes)
path = "D:/ziliao/R语言 学习/R语言构建单倍型网络"
setwd(path)
x<-read.fas(file="DatasetB03.fas")
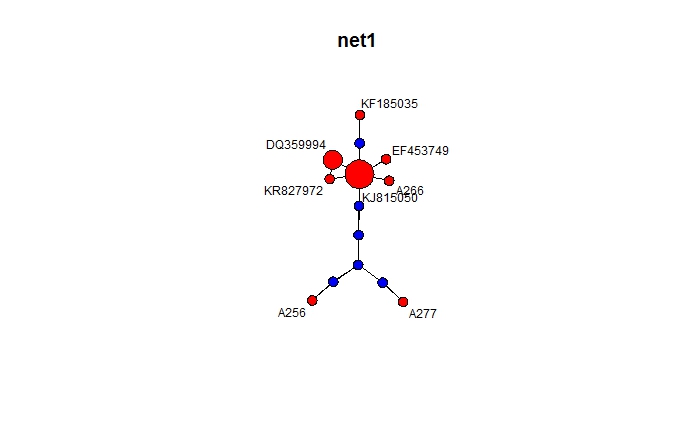
p<-parsimnet(x,prob=.97) #设定遗传距离阈值分割网络
plot(p,net=1)
plot(p,net=2)
plot(p,net=3)
plot(p,net=4)
plot(p,net=5)
plot(p,net=6)
#plot(p, interactive=TRUE,vertex.cex=c(rep(3,nrow(p@d[[1]]))))
plot(p,interactive=TRUE,vertex.cex=c(2, 2, 2, 4, 2, 2, 6, 2, 2, 2, 2, 2, 2, 2)) #逐个试试具体哪个单倍型排在什么位置,设置相应大小
h<-haplotype(x,indels="sic")
h #查看单倍型分类情况
https://m.sciencenet.cn/blog-508298-1069194.html
上一篇:Mrbayes出错原因
下一篇:如何解读TCS单倍型网络图