博文
MMseqs2软件cluster(聚类)
|

软件安装
conda安装
1. conda create -n mmseqs2 2. conda activate mmseqs2 3. conda install -c https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/conda-forge/ -c https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/bioconda/ mmseqs2 |

聚类分析
1. easy-cluster (核心算法:cascaded clustering algorithm)
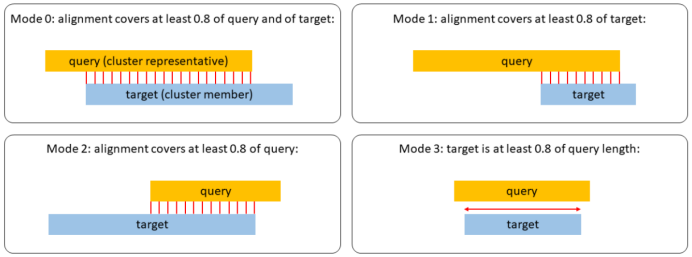
mmseqs easy-cluster examples.fasta clusterRes tmp --min-seq-id 0.5 -c 0.8 --cov-mode 1 结果解释: clusterRes 结果文件的前缀。 tmp生成tmp/文件夹,里面记录每次的分析过程。 --min-seq-id 相似性阈值 -c 覆盖度阈值 --cov-mode coverage模式(详见下图) 0 :bidirectional 1 :target coverage 2 :query coverage 3 :target-in-query length coverage |
2. easy-linclust(适合大数据集)
mmseqs easy-linclust examples.fasta clusterRes tmp |
备注:
1. --cluster-mode 聚类模型参数,默认是2(2:Greedy clustering by sequence length (CDHIT))
2. 推荐参数组合 --cluster-mode 2 --cov-mode 1

结果解释
⑴
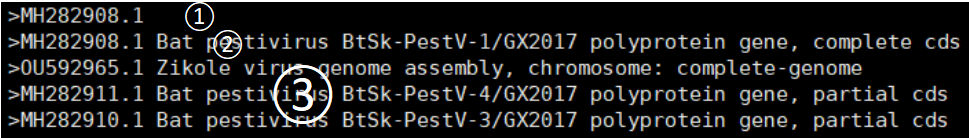
*_all_seqs.fasta(总聚类序列文件,分3部分)
1.该聚类名称(代表性序列的seqid号);2.该聚类的代表性序列;3.该聚类的非代表性序列。
⑵
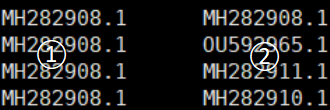
*_cluster.tsv (聚类结果清单,和*_all_seqs.fasta一致。分2部分)
1. 第一列为cluster-representative(聚类的代表性序列seqid号);2. 第二列为cluster-member(聚类的序列成员seqid号)。
⑶
*_rep_seq.fasta (所有的representative sequences,聚类的代表性序列)

⑷
tmp/ 文件夹里面记录每次的分析过程
【参考】
【1】https://github.com/soedinglab/MMseqs2
https://m.sciencenet.cn/blog-994715-1391228.html
上一篇:第二代测序技术原理精讲
下一篇:seqkit软件用法小记