博文
《自然—方法》:清华大学戴琼海/俞立/吴嘉敏合作开发自监督显微图像增强方法,助力细胞生物学新发现
||
荧光显微镜已成为揭示细胞和细胞器动态调控的不可或缺的工具。然而,固有的成像噪声限制了光学显微的成像质量。如何平衡高帧率、长时间、低光毒性、高保真度是细胞生物学成像的基础难题。
北京时间2023年11月14日,清华大学戴琼海、俞立和吴嘉敏等合作在《自然—方法》(Nature Methods)发表题为“Bio-friendly long-term subcellular dynamic recording by self-supervised image enhancement microscopy”的论文。 研究团队提出了DeepSeMi,一种基于自监督学习的图像增强框架。通过引入新设计的盲点神经网络,DeepSeMi可以有效地增强显微图像,而不会损失时空分辨率。结合共聚焦显微镜,DeepSeMi以高帧率记录四种荧光标记的细胞器互作,记录迁移体和收缩丝体跨越数万帧下的形态变化。同时,使用DeepSeMi可以在共聚焦显微镜下观测光毒性非常敏感的盘基网柄菌。 通过对各种样品和仪器进行全面验证,研究人员证明了DeepSeMi是一种多功能且生物相容的工具,能够突破光子噪声限制。

DeepSeMi的创新源于对噪声统计的充分利用。由于图像局部的连续性,甚在不包括目标像素本身的情况下,从领域像素到目标像素之间建立良好的映射。基于这一观察,DeepSeMi通过盲点卷积神经网络,有效地建立了噪声图像中邻域像素与目标像素之间的映射关系,以实现图像增强。
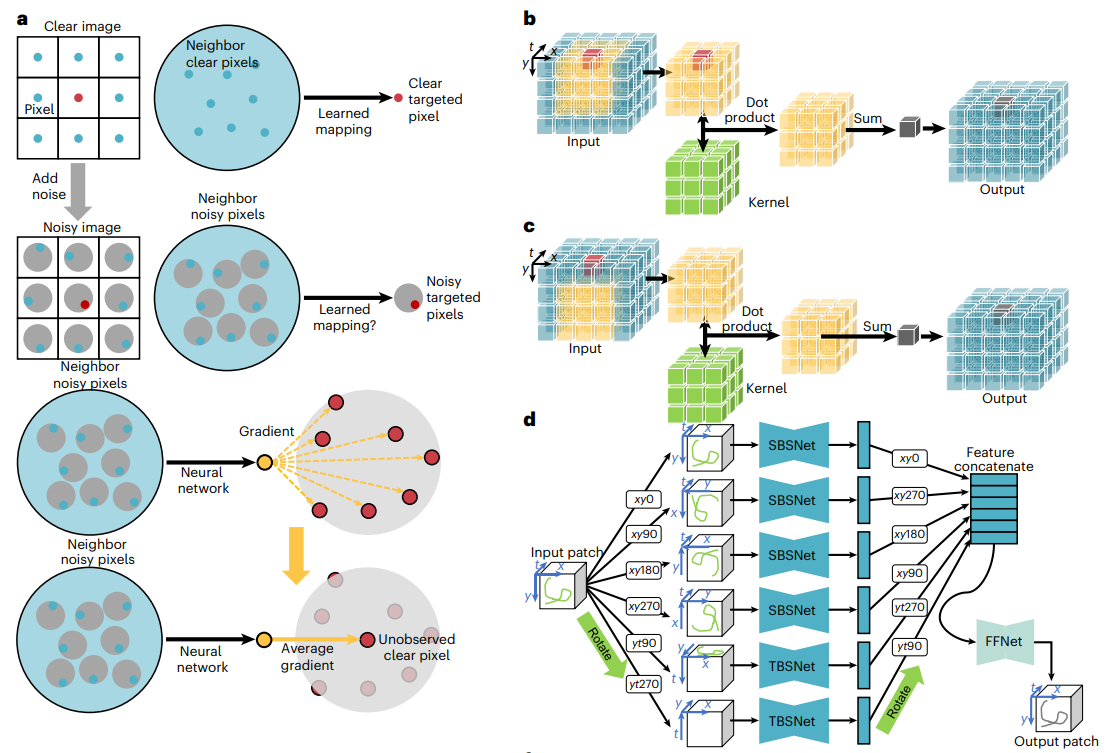
图1. DeepSeMi的基本架构
通过DeepSeMi,研究人员以2帧/秒的高分辨率实现了跨越在半天的时间下的迁移体生成、膨胀和破裂过程成像,在超低光强下共采集了超过86,000个时间点(488nm为45.3µW,561nm为49.8µW)。DeepSeMi增强后的成像结果清楚地显示了一些线粒体被细胞排出并保留在迁移体内的情况,这被称为mitocytosis。原始图像中噪声完全将迁移体的形态淹没,研究人员从整个DeepSeMi增强的结果中中分割出了51个迁移体,并实现了迁移体面积随时间的变化进行统计。收缩丝体作为一种新型的小型细胞外囊泡,它是由收缩丝生成,并与细胞迁移密切相关。传统的共聚焦成像受限于光毒性,无法对收缩丝体的形成过程进行完整记录,而所提出的DeepSeMi能够将收缩丝体生成过程清晰地恢复出来。
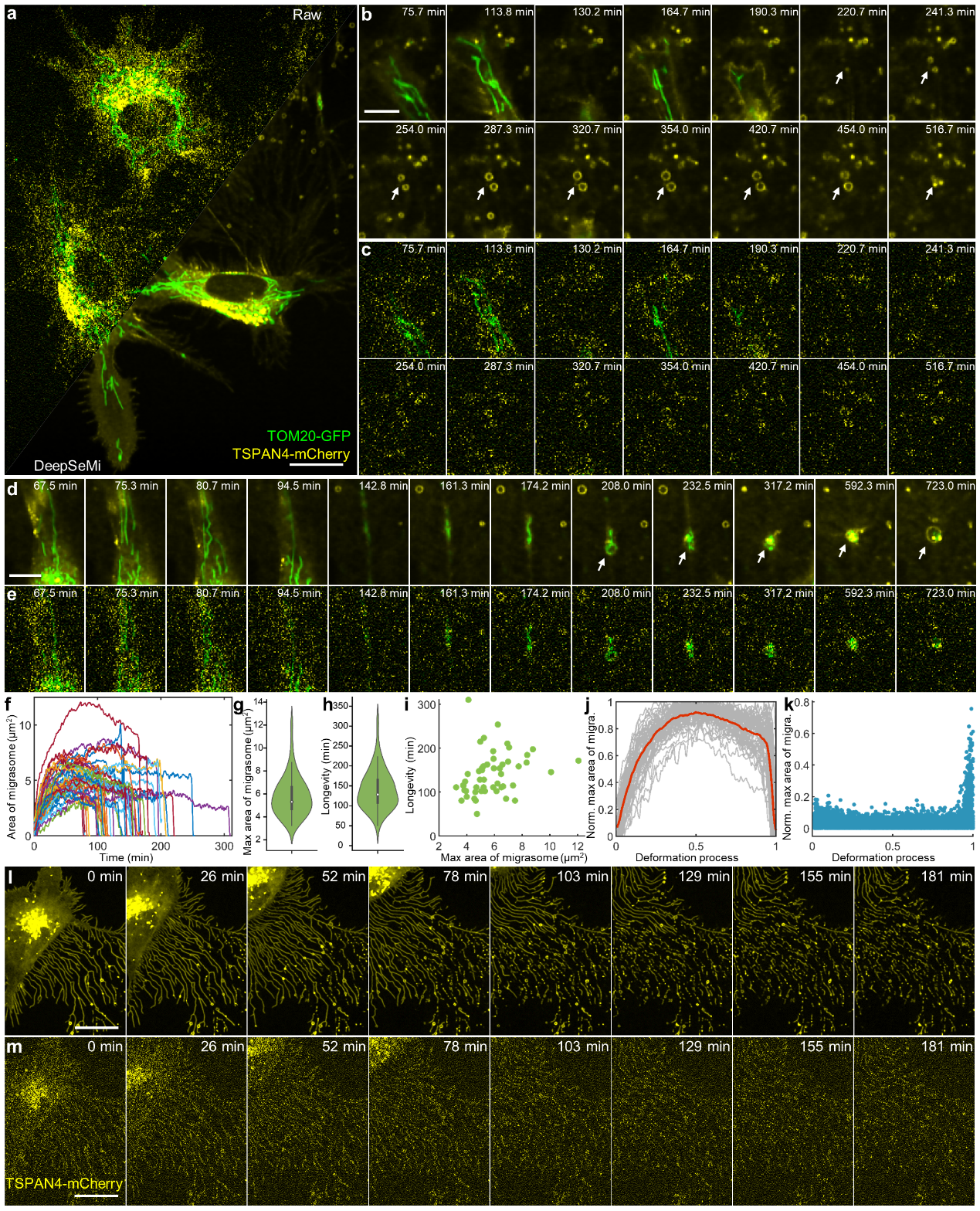
图2. DeepSeMi用于迁移体和伸缩丝体形成过程的活细胞成像
DeepSeMi增强后的成像结果也有助于细胞和细胞器的自动分析,有助于处理大规模成像数据。此外,DeepSeMi在线虫、斑马鱼和小鼠等各种生物样本上也有优异的性能。综合而言,DeepSeMi提供了高分辨率、高速度、多色成像和低光漂白、光毒性的组合,非常适合细胞生物学的科学研究。
研究团队长期深耕智能增强显微成像领域,提出了多种性能优越且用户友好的自监督显微图像增强方法。2021年,团队在期刊Nature Methods上发表面向双光子钙成像的时空增强方法DeepCAD;2022年,团队在期刊Nature Biotechnology上发表DeepCAD的综合优化版本DeepCAD-RT,对DeepCAD进行了网络结构简化和硬件部署优化,能够在成像过程中实时对采集结果进行处理;DeepSeMi是团队继续拓宽“Denoise”宇宙,面向细胞生物学设计的显微成像增强新方法。目前团队正在将这些方法进行整体集成从而形成“一键式”运行的软件,更加便于生命科学研究者的部署和使用。
清华大学自动化系博士张国勋、清华大学生命学院博士李晓鹏、清华大学自动化系助理研究员张元龙为该文章的共同第一作者;清华大学脑与认知科学研究院、清华大学自动化系戴琼海教授,清华大学生命学院俞立教授、清华大学自动化系吴嘉敏副教授为该论文共同通讯作者。
相关论文信息:
https://doi.org/10.1038/s41592-023-02058-9
参考文献
1. Li, X. et al. Real-time denoising enables high-sensitivity fluorescence time-lapse imaging beyond the shot-noise limit. Nat. Biotechnol. (2022), doi: 10.1038/s41587-022-01450-8
2. Li, X. et al. Reinforcing neuron extraction and spike inference in calcium imaging using deep self-supervised denoising. Nat. Methods 18, 1395-1400, doi:10.1038/s41592-021-01225-0 (2021).
https://m.sciencenet.cn/blog-3423233-1410100.html
上一篇:《细胞》: 福建农林大学徐通达/杨贞标合作揭示植物激素生长素胞外新受体
下一篇:《自然》: 陈靖/何川/迟洪波合作揭示食物来源血液营养物在抗病免疫中的重要作用